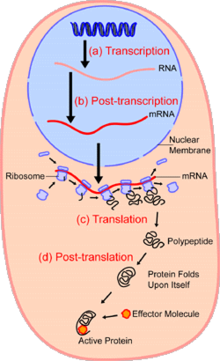
단세포 분석
세포생물학에서 단세포 분석은 단일 세포 수준에서 유전체학, 전사체학, 단백질체학, 대사체학 및 세포 간 상호작용에 대한 연구이다.[1][2][3] 진핵 및 원핵 세포군 모두에서 이질성(heterogeneity)이 보여지므로, 단일 세포를 분석하면 대량의 세포 집단을 연구할 때 볼 수 없는 메커니즘을 발견할 수 있다.[4] FACS(fluorescence-activated cell sorting)와 같은 기술을 사용하면 시료에서 특정 단일 세포를 정확하게 분리할 수 있다. 이는 처리량이 많은 단일 세포 분할 기술[5][6][7]을 통해 동시에 수백 또는 수천 개의 단일 세포의 분자 분석이 가능하다; 이는 특히 유전자형이 동일한 세포에서 전사체 변이를 분석하는 데 유용하며, 그 외 감지되지 않는 세포 하위 유형을 정의할 수 있게 된다. 새로운 기술의 개발은 단일 세포의 게놈과 전사체를 분석하고[8] 단백질체와 대사체를 정량하는 능력을 향상 시키고 있다.[9][10][11] 질량 분석 기술은 단일 세포의 단백질체 및 대사체 분석을 위한 중요한 분석 기법이 되었다.[12][13] 최근 발전으로 수백 개의 단일 세포에서 수천 개의 단백질 정량이 가능하게 되었고[14], 이로 인해 새로운 유형의 분석이 가능해졌다.[15][16] In situ sequencing 및 형광 원위치 혼성화 (Fluorescence in situ hybridzation, FISH)는 세포를 분리하지 않아도 되며 조직 분석에 점점 더 많이 사용되고 있다.[17]
단일 세포 분리 편집
많은 단일 세포 분석 기술은 각 세포의 분리를 필요로 한다. 현재 단일 세포 분리에 사용되는 방법으로 : 유전영동 디지털 분류(dielectrophoretic digital sorting), FACS, 동수집유장(hydrodynamic traps), 레이저 포착 현미해부(laser capture microdissection), 미세 유체 공학(microfluidics), 현미조작(micromanipulation), 연속희석법(serial dilution) 등이 있다.
관련 기술 편집
유전영동 디지털 분류법은 미세유체칩의 반도체 제어 전극을 활용하여 유전영동(DEP) 케이지에서 단일 세포를 포획하는 기법으로, 세포의 식별은 형광 마커의 이미지 관찰을 통해 검증될 수 있다. 또한, 유세포에서 DEP 케이지의 반도체 제어 동작을 통해 정확히 전달되었는지 확인할 수 있다.
유체역학적 포획은 수동적 미세유체 수송에 의해 각각 주어진 시간에 "트랩"에 각 세포를 분리할 수 있다. 분리된 세포의 수는 시스템의 트랩 수에 기반하여 조작할 수 있다.
레이저 포착 현미해부 기술은 보고자 하는 조직 샘플에서 개체 세포 또는 특정 부분을 해부하고 분리하기 위해 레이저를 사용한다. 이 방법은 현미경으로 세포를 관찰하여 분석할 부분을 식별하고 표지하여 레이저로 세포를 절단하게 된다. 그런 다음 분석을 위해 세포를 추출한다.
수동 단일 세포 선택은 세포를 현미경으로 관찰하고 마이크로피펫 을 사용하여 선택하는 방법이다.[18][19]
미세 유체 공학은 추후 분석을 위한 개별 세포의 분리에 사용된다. 다음 원칙은 단일 세포의 분리를 위한 다양한 미세 유체 과정을 간략하게 설명하고 있다: droplet-in-oil 기반 분리, 공기막 밸브 및 유체 역학 세포 트랩. Droplet-in-oil 기반 미세 유체는 분리된 aqueous droplet을 유지하기 위해 오일로 채워진 채널을 사용한다. 이를 통해서 단일 세포는 오일 기반 채널 내부에 들어가 분리될 수 있다. 공압 멤브레인 밸브는 멤브레인 편향에 의한 개별 세포의 분리를 위해 공기압 조작을 사용한다. 압력의 조절은 미세 유체 네트워크에서 채널을 열거나 닫을 수 있도록 한다. 일반적으로 이 시스템은 운영자가 필요하며 제한된 처리량을 가지고 있다.
라만 핀셋 기술은 라만 분광법과 광학 핀셋이 결합된 기법으로 레이저 빔을 사용하여 세포를 포획하고 조작하는 데 사용된다.[20]
유체역학 기반 미세유체 바이오칩의 개발은 수년에 걸쳐 증가하고 있다. 이 기술에서 세포는 단일 세포 분석(SCA)을 위해 특정 영역에 갇혀 있게 된다. 이는 일반적으로 광학, 전기, 자기 또는 음향과 같은 외부 힘 필드의 적용 없이 발생한다. 세포의 자연 상태에서 SCA에 대한 통찰력을 탐구할 필요가 있으며 이러한 기술의 개발은 해당 연구에 매우 중요하다. 연구원들은 시장 및 연구원 요구에 맞는 바이오칩 장치를 개발할 필요성을 강조하고 있으며, 유체역학적 미세유체학은 수동적인 랩온칩의 개발을 용이하게 한다.
유전체학 편집
기술 편집
단일 세포 유전체학은 세포에서 발견되는 DNA 사본의 증가에 크게 의존하므로 염기서열 분석에 충분하다. 이는 전체 게놈 증폭 (WGA)을 위한 전략의 개발로 이어졌다. 현재 WGA 전략은 세 가지 범주로 나뉘어진다.
- 제어된 프라이밍 및 PCR 증폭: 어댑터-링커 PCR WGA
- 무작위 프라이밍 및 PCR 증폭: DOP-PCR, MALBAC
- 무작위 프라이밍 및 등온 증폭: MDA
Adapter Linker PCR WGA는 aCGH와 NGS 저역 통과 시퀀싱과 함께 매우 낮은 Allelic Dropout 효과로 인해 이배체 단일 세포 돌연변이 분석[21][22][23] 그리고 낮은 노이즈로 인해 복제수 변이 프로파일링에서 가장 우수한 성능을 보이는 것으로 많은 비교 연구를 통해 보고되었다.[24][25] 이 방법은 고정 및 고정되지 않은 인간 세포에만 적용할 수 있다.
널리 채택된 WGA 기술 중 하나는 축중 올리고뉴클레오티드 기반 중합효소연쇄반응(DOP-PCR)으로, 이는 잘 확립된 DNA 증폭 방법인 PCR을 사용하여 많은 프라이머 조합을 사용하여 전체 게놈을 증폭한다. 이 방법은 간단하지만 게놈 적용 범위가 매우 낮은 것으로 나타났다. 이에 따라, DOP-PCR는 무작위 프라이머 및 Φ29 DNA 중합효소와 같이 높은 정확도를 가지는 효소를 사용하여 다중 변위 증폭 (MDA)을 통해 보다 더 큰 단편과 더 큰 게놈 범위의 증폭이 가능하도록 개선되어야 한다. 이러한 개선에도 MDA는 여전히 서열 의존적 편향을 보인다 (서열로 인해 게놈의 특정 부분이 다른 부분보다 더 많이 증폭된다). DOP-PCR 및 MDA에서 볼 수 있는 편향을 크게 피하기 위해 표시된 방법은 다중 결합 및 루프 기반 증폭 주기 (MALBAC)이다. 이 시스템의 편향은 DNA 사본의 사본을 만드는 대신 원래의 DNA 가닥만 복사함으로써 감소된다. MALBA 사용의 주요 단점은 DNA를 복사하는 데 사용되는 효소로 인해 DOP-PCR 및 MDA에 비해 정확도가 떨어진다는 것이다.[9] 위의 기술 중 하나를 사용하여 증폭된 DNA 가닥은 Sanger 또는 NGS( 차세대 시퀀싱)를 사용하여 시퀀싱할 수 있다.
목적 편집
단일 세포 수준에서 게놈을 연구하는 데에는 두 가지 주요 활용 방법이 있다. 하나는 표현형 차이가 자주 보이는 박테리아 개체군에서 발생하는 변화를 추적하는 것이다. 이러한 차이는 집단에서의 대량 시퀀싱에서는 놓칠 수 있지만 단일 세포 시퀀싱에서는 관찰이 가능하다.[26] 두 번째는 활용 방법은 암의 유전적 진화를 연구하는 것이다. 암세포는 끊임없이 돌연변이를 일으키기 때문에 암이 유전적 수준에서 어떻게 진화하는지 보는 것은 관심을 가지기 매우 충분하다. 이때 나타나는 체세포 돌연변이 및 복제 수 이상 패턴은 단일 세포 시퀀싱을 사용하여 관찰할 수 있다.[1]
전사체학 편집
기술 편집
단일 세포 전사체는 단일 세포 게놈과 유사한 시퀀싱 기술을 사용하거나 형광 제자리 혼성화를 통한 직접 검출을 사용한다. 전사체를 정량하기 위한 첫 번째 단계는 역전사 효소를 사용하여 RNA로 cDNA을 합성하여 유전체학에서 수행된 것처럼 NGS 방법을 통해 세포의 정보를 시퀀싱할 수 있도록 하는 것이다. 한 번 합성되면, 시퀀싱을 위한 cDNA가 충분하지 않으므로, 단일 세포 유전체학에서 논의된 동일한 DNA 증폭 기술이 cDNA에 적용된다.[1] 혹은, RNA 혼성화 probe에 부착된 형광 물질을 사용하여 특정 서열을 식별하고 다양한 RNA probe를 순차적으로 적용하여 포괄적인 전사체를 구축할 수 있다.[27][28]
목적 편집
단일 세포 전사체의 목적은 각 세포에서 어떤 유전자가 발현되는지 확인하는 것이다. 전사체는 단백질 수준에서 증폭이 어렵기 때문에 단백질체 대신 유전자 발현을 정량화하는 데 종종 사용된다.[1]
이 기술을 사용하여 유전자 발현을 연구하는 데에는 세 가지 이유가 있다. 이는 유전자 역학, RNA 스플라이싱 및 세포 유형을 연구하기 위함이다. 유전자 역학은 일반적으로 어떠한 유전자 발현의 변화가 다른 세포 특성에 영향을 미치는지 확인하기 위해 연구된다. 예를 들어, 이러한 유형의 전사체 분석은 종종 배아 발달을 연구하는 데 사용되었다. RNA 스플라이싱 연구는 다양한 전사체 이소폼의 조절을 이해하는 데 중점을 두고 있다. 또한, 단일 세포 전사체는 세포 유형화에 사용되어 왔으며, 세포에서 발현된 유전자는 세포 유형을 식별하는 데 사용된다. 세포 유형화의 주요 목표는 알려진 유전 표지가 없는 세포의 정체를 확인하는 방법을 찾는 것이다.[1]
단백질체학 편집
기술 편집
단일 세포 단백질체학에는 항체 기반 방법, 형광 단백질 기반 방법 및 질량 분광법 기반 방법의 세 가지 주요 접근 방식이 있다.[29][16]
항체 기반 방법 편집
항체 기반 방법은 관심 단백질에 결합하기 위해 설계된 항체를 사용하며, 여러 다른 기술 중 하나를 사용해 여러 개별 표적을 상대적으로 많이 식별할 수 있다.
이미징 : 항체는 양자점 또는 유기 형광단으로 표지된 같은 형광 분자에 결합하여 형광 현미경으로 검출될 수 있다. 서로 다른 색의 양자점 또는 특정 형광단이 각 항체에 부착되어 있기 때문에 단일 세포에서 여러 개의 서로 다른 단백질을 식별할 수 있다. 양자점은 샘플을 손상시키지 않고 항체에서 씻어낼 수 있으므로, 동일한 샘플에서 이 방법을 사용하여 단백질 정량을 여러 번 수행할 수 있다.[30] 유기 형광단을 기반으로 하는 방법의 경우, DNA-혼성과 같이 가역적 연결에 의해 부착되거나 (저염 조건에서 녹거나 해리될 수 있다)[31] 화학적으로 비활성화되어 여러 번의 분석이 가능한[32] 형광 표지를 사용하여 각 주기당 3-5개의 표적을 정량화한다. 이러한 접근법은 환자 생검 샘플(예: 암)에서 풍부한 단백질을 정량하여 조직 그리고/또는 종양에서 가변 단백질 발현을 표시하고[32] 암 치료에 대한 반응으로 단백질 발현 및 세포 신호전달의 변화를 측정하기 위해 사용되었다.[31]
질량 세포분석 : 세포나 조직에서 흔히 발견되지 않는 희소 금속 동위원소를 개별 항체에 부착하여 질량분석기로 단백질을 동시에 검출하고 민감하게 식별할 수 있다.[33] 이러한 기술은 단일 세포에서 많은 표적 (최대 38개)를 동시에 정량화하기 위해 다중화될 수 있다.[34]
항체-DNA 정량화 : 또 다른 항체 기반 방법은 단백질 수준을 DNA 수준으로 변환하는 것이다.[29] DNA로의 전환은 단백질 수준을 증폭하고 NGS를 사용하여 단백질을 정량화하는 것을 가능하게 한다. 하나의 접근 방식으로, 정량화해야 하는 각 단백질에 대해 2개의 항체가 선택된다. 그럼 다음 두 항체는 상보적인 단일 가닥 DNA가 연결되도록 수정된다. 두 개의 항체가 단백질에 결합하면 상보적인 가닥이 결합되어 DNA의 이중 가닥 부분이 생성되고 PCR을 통해 증폭될 수 있다. 한 단백질에 대해 설계된 각 항체 쌍은 다른 DNA 서열로 표지된다. 그런 다음 PCR을 통해 증폭된 DNA를 시퀀싱하고 단백질 수준에서 정량할 수 있다.[35]
질량 분광법 기반 방법 편집
질량 분광법 기반 단백질체학에서 펩타이드 식별을 위한 세 가지 주요 단계가 있다: 샘플 준비, 펩타이드 분리 및 펩타이드 식별. 몇몇 그룹은 비정상적으로 크고 분석에 충분한 물질을 제공할 수 있는 난모세포 또는 초기 분열 단계 세포에 초점을 맞추었다.[36][37][38] 또 다른 접근 방식으로, 질량 분석법에 의한 단일 세포 단백질체학(SCoPE-MS)은 운반체 세포와 단일 세포 바코드를 결합하여 포유동물 세포에서 전형적인 세포 크기(직경 10-15μm)를 가진 수천 개의 단백질을 정량화했다.[39][40][41] 2세대 SCoPE-MS인 SCoPE2는 자동화 그리고 샘플 준비의 간결화를 통해 처리량을 증가시켰다; 하나의 접근 방법은 MAMS(질량 분석을 위한 마이크로어레이)로, recipient site와 주변 영역 간의 습윤성의 차이를 이용하여 빠른 속도로 부분 표본을 할 수 있다.[42] 또한, LC-MS/MS 및 펩타이드 식별의 데이터 기반 최적화를 통해 정량적 신뢰성과 단백질체학 범위를 개선하였다. 분석을 위해 펩타이드를 분리하는 여러 방법이 있는데, 여기에는 필터 지원 샘플 준비 (FASP), 자기 비드 사용 또는 일련의 시약 및 원심 분리 단계의 사용이 포함된다.[43][36][38] 서로 다른 크기를 가진 단백질의 분리는 모세관 전기영동 (CE) 또는 액체 크로마토그래피 (LC)를 사용하여 수행할 수 있다 (질량 분광법과 함께 액체 크로마토그래피를 사용하는 것은 LC-MS라고도 한다).[36][37][38] 이 단계는 탠덤 질량 분광법 (MS/MS)을 통한 단백질의 정량화 전에 펩타이드에 순서를 부여한다. 정량화 방법에 있어 주요 차이점은 일부는 펩타이드에 탠덤 질량 표지(TMT) 또는 디메틸 라벨과 같은 라벨을 사용하여 특정 단백질이 어느 세포에서 유래했는지(각 세포에서 유래한 단백질은 다른 라벨을 가짐) 식별하는 데 사용되는 데 반면, 다른 것들은 라벨을 사용하지 않고 개별적으로 세포를 정량화한다는 것이다. 질량 분석 데이터는 식별된 펩타이드에 대한 정보를 단백질 수준의 정량화로 변환하는 데이터베이스를 통해 실행되어 분석된다.[36][37][38][44]
목적 편집
단백질체를 연구하는 목적은 단일 세포 수준에서 세포의 활동을 보다 더 이해하는 것이다. 단백질은 세포가 어떻게 작용하는지 결정하는 역할을 하므로 단일 세포의 단백질체에 대한 이해는 세포가 작동하는 방식과 다양한 환경 자극으로 인해 세포에서 유전자 발현이 어떻게 변화하는지 이해할 수 있도록 도와준다. 전사체학은 단백질체학과 같은 목적을 가지고 있지만 전사 후 조절 을 고려하지 않기 때문에 세포의 유전자 발현을 결정하는 데에는 정확하지 않다.[10] RNA 수준과 단백질 수준의 차이를 연구하면 전사 후 조절되는 유전자에 대한 통찰력을 얻을 수 있기 때문에 전사체학은 여전히 중요하다.
대사체학 편집
기술 편집
단일 세포의 대사체를 정량화하는 데 사용되는 네 가지의 주요 방법이 있으며, 이는 형광 기반 검출, 형광 바이오센서, FRET 바이오센서 및 질량 분광법이다. 앞에 나열된 세 가지 방법은 형광 현미경을 사용하여 세포의 분자를 확인한다. 일반적으로 이러한 분석은 관심 분자에 부착된 작은 형광 표지를 사용하지만, 이는 단일 세포 대사체학에는 너무 침습적이며 대사 산물의 활성을 변형시킬 수도 있다. 이 문제에 대한 해결 방법으로 관심 대사 산물에 결합할 때 형광을 발하는 대사 산물 검출기 역할을 하는 형광 단백질을 사용하는 것이다.[45]
질량 분광법은 단일 세포 대사체학에 가장 자주 사용되는 방법이 되고 있다. 이 방법의 장점은 모든 관심 분자에 대한 형광 단백질을 개발할 필요가 없고 펨토몰 범위의 대사 산물까지 감지할 수 있다는 것이다.[13] 단백질체학에서 논의된 방법과 유사하게, 질량 분광법을 모세관 전기영동법과 같은 분리 기술과 결합하여 대사 산물을 정량할 수 있었다. 이 방법은 또한 펨토몰 농도에 존재하는 대사 산물을 검출할 수 있다.[45] 모세관 마이크로샘플링을 활용하는 또 다른 방법은 이온 이동성 분리가 있는 질량 분광법과 결합하여 단일 세포 대사체학에 대한 분자 범위 및 이온 분리를 향상시키는 것으로 입증되었다.[19][46] 연구원들은 현재 기술의 부족한 점을 충족시킬 수 있는 기술을 개발하기 위해 노력하고 있다: 높은 처리량, 풍부하지 않거나 낮은 이온화 효율을 가지는 대사 산물에 대한 더 높은 민감도, 우수한 재현성, 대사 산물의 정량화.[47]
목적 편집
단일 세포 대사체학의 목적은 암, 줄기 세포, 노화 및 약물 내성 발달에 대해 분자 수준에서 더 나은 이해를 얻는 것이다. 일반적으로 대사체학의 초점은 대부분 분자 수준에서 세포가 환경 스트레스에 반응하는 방식을 이해하고 세포 기능에 대한 보다 역동적인 이해를 제공하는 것이다.[45]
세포 간 상호작용 편집
세포 간 상호작용은 안정적이고 일시적인 상호작용이다.
각주 편집
- ↑ 가 나 다 라 마 “Single cell analysis: the new frontier in 'omics'”. 《Trends in Biotechnology》 28 (6): 281–90. June 2010. doi:10.1016/j.tibtech.2010.03.002. PMC 2876223. PMID 20434785.
- ↑ “Computation and measurement of cell decision making errors using single cell data”. 《PLOS Computational Biology》 13 (4): e1005436. April 2017. Bibcode:2017PLSCB..13E5436H. doi:10.1371/journal.pcbi.1005436. PMC 5397092. PMID 28379950.
- ↑ “Automated profiling of individual cell-cell interactions from high-throughput time-lapse imaging microscopy in nanowell grids (TIMING)”. 《Bioinformatics》 31 (19): 3189–97. October 2015. doi:10.1093/bioinformatics/btv355. PMC 4693004. PMID 26059718.
- ↑ “Cellular heterogeneity: do differences make a difference?”. 《Cell》 141 (4): 559–63. May 2010. doi:10.1016/j.cell.2010.04.033. PMC 2918286. PMID 20478246.
- ↑ “Single Cell Isolation and Analysis”. 《Frontiers in Cell and Developmental Biology》 4: 116. 2016년 10월 25일. doi:10.3389/fcell.2016.00116. PMC 5078503. PMID 27826548.
- ↑ “Miniaturization Technologies for Efficient Single-Cell Library Preparation for Next-Generation Sequencing”. 《Journal of Laboratory Automation》 21 (4): 557–67. August 2016. doi:10.1177/2211068216630741. PMC 4948133. PMID 26891732.
- ↑ “Massively parallel digital transcriptional profiling of single cells”. 《Nature Communications》 8: 14049. January 2017. Bibcode:2017NatCo...814049Z. doi:10.1038/ncomms14049. PMC 5241818. PMID 28091601.
- ↑ “Single-Cell Gene Network Analysis and Transcriptional Landscape of MYCN-Amplified Neuroblastoma Cell Lines”. 《Biomolecules》 11 (2): 177. January 2021. doi:10.3390/biom11020177. PMC 7912277. PMID 33525507.
- ↑ 가 나 “Single-Cell Whole-Genome Amplification and Sequencing: Methodology and Applications”. 《Annual Review of Genomics and Human Genetics》 16 (1): 79–102. 2015. doi:10.1146/annurev-genom-090413-025352. PMID 26077818.
- ↑ 가 나 “Single-Cell Transcriptional Analysis”. 《Annual Review of Analytical Chemistry》 10 (1): 439–462. June 2017. doi:10.1146/annurev-anchem-061516-045228. PMID 28301747.
- ↑ “A new toolbox for assessing single cells”. 《Annual Review of Chemical and Biomolecular Engineering》 5: 455–77. 2014. doi:10.1146/annurev-chembioeng-060713-035958. PMC 4309009. PMID 24910919.
- ↑ “Categorizing Cells on the Basis of their Chemical Profiles: Progress in Single-Cell Mass Spectrometry”. 《Journal of the American Chemical Society》 139 (11): 3920–3929. March 2017. doi:10.1021/jacs.6b12822. PMC 5364434. PMID 28135079.
- ↑ 가 나 “Single-Cell Mass Spectrometry Approaches to Explore Cellular Heterogeneity”. 《Angewandte Chemie》 57 (17): 4466–4477. April 2018. doi:10.1002/anie.201709719. PMID 29218763.
- ↑ “Single-cell protein analysis by mass spectrometry”. 《Current Opinion in Chemical Biology》 60: 1–9. June 2020. arXiv:2004.02069. doi:10.1016/j.cbpa.2020.04.018. ISSN 1367-5931. PMC 7767890. PMID 32599342.
- ↑ “Transformative Opportunities for Single-Cell Proteomics”. 《Journal of Proteome Research》 17 (8): 2565–2571. August 2018. doi:10.1021/acs.jproteome.8b00257. PMC 6089608. PMID 29945450.
- ↑ 가 나 “Unpicking the proteome in single cells”. 《Science》 367 (6477): 512–513. January 2020. Bibcode:2020Sci...367..512S. doi:10.1126/science.aaz6695. PMC 7029782. PMID 32001644.
- ↑ “De Novo Gene Expression Reconstruction in Space”. 《Trends in Molecular Medicine》 23 (7): 583–593. July 2017. doi:10.1016/j.molmed.2017.05.004. PMC 5514424. PMID 28571832.
- ↑ “Technologies for Single-Cell Isolation”. 《International Journal of Molecular Sciences》 16 (8): 16897–919. July 2015. doi:10.3390/ijms160816897. PMC 4581176. PMID 26213926.
- ↑ 가 나 “Energy Charge, Redox State, and Metabolite Turnover in Single Human Hepatocytes Revealed by Capillary Microsampling Mass Spectrometry”. 《Analytical Chemistry》 87 (20): 10397–405. October 2015. doi:10.1021/acs.analchem.5b02502. PMID 26398405.
- ↑ 〈Analysis of single eukaryotic cells using Raman Tweezers〉. 《Single-Cell Analysis》. Methods in Molecular Biology 853. Humana Press. January 2012. 151–67쪽. doi:10.1007/978-1-61779-567-1_12. ISBN 978-1-61779-566-4. PMID 22323146.
- ↑ “Comparative study of whole genome amplification and next generation sequencing performance of single cancer cells”. 《Oncotarget》 8 (34): 56066–56080. August 2017. doi:10.18632/oncotarget.10701. PMC 5593545. PMID 28915574.
- ↑ “A new workflow for whole-genome sequencing of single human cells”. 《Human Mutation》 35 (10): 1260–70. October 2014. doi:10.1002/humu.22625. PMID 25066732.
- ↑ “Comparison of whole genome amplification techniques for human single cell exome sequencing”. 《PLOS ONE》 12 (2): e0171566. 2017. Bibcode:2017PLoSO..1271566B. doi:10.1371/journal.pone.0171566. PMC 5313163. PMID 28207771.
- ↑ “Comparison of three whole genome amplification methods for detection of genomic aberrations in single cells”. 《Prenatal Diagnosis》 36 (9): 823–30. September 2016. doi:10.1002/pd.4866. PMID 27368744.
- ↑ “STR profiling and Copy Number Variation analysis on single, preserved cells using current Whole Genome Amplification methods”. 《Scientific Reports》 7 (1): 17189. December 2017. Bibcode:2017NatSR...717189V. doi:10.1038/s41598-017-17525-5. PMC 5719346. PMID 29215049.
- ↑ “Single-cell genomics”. 《Nature Methods》 8 (4): 311–4. April 2011. doi:10.1038/nmeth0411-311. PMID 21451520.
- ↑ “Single-cell in situ RNA profiling by sequential hybridization”. 《Nature Methods》 11 (4): 360–1. April 2014. doi:10.1038/nmeth.2892. PMC 4085791. PMID 24681720.
- ↑ “RNA imaging. Spatially resolved, highly multiplexed RNA profiling in single cells” (PDF). 《Science》 348 (6233): aaa6090. April 2015. doi:10.1126/science.aaa6090. PMC 4662681. PMID 25858977.
- ↑ 가 나 “Single cell protein analysis for systems biology”. 《Essays in Biochemistry》 62 (4): 595–605. October 2018. doi:10.1042/EBC20180014. PMC 6204083. PMID 30072488.
- ↑ “Multicolor multicycle molecular profiling with quantum dots for single-cell analysis”. 《Nature Protocols》 8 (10): 1852–69. October 2013. doi:10.1038/nprot.2013.112. PMC 4108347. PMID 24008381.
- ↑ 가 나 “Single-cell barcode analysis provides a rapid readout of cellular signaling pathways in clinical specimens”. 《Nature Communications》 9 (1): 4550. October 2018. Bibcode:2018NatCo...9.4550G. doi:10.1038/s41467-018-07002-6. PMC 6208406. PMID 30382095.
- ↑ 가 나 “Highly multiplexed immunofluorescence imaging of human tissues and tumors using t-CyCIF and conventional optical microscopes”. 《eLife》 7: e31657. July 2018. doi:10.7554/eLife.31657. PMC 6075866. PMID 29993362.
- ↑ “Mass cytometry as a platform for the discovery of cellular biomarkers to guide effective rheumatic disease therapy”. 《Arthritis Research & Therapy》 17: 127. May 2015. doi:10.1186/s13075-015-0644-z. PMC 4436107. PMID 25981462.
- ↑ “Mass Cytometry: Single Cells, Many Features”. 《Cell》 (영어) 165 (4): 780–91. May 2016. doi:10.1016/j.cell.2016.04.019. PMC 4860251. PMID 27153492.
- ↑ “Simple Method To Prepare Oligonucleotide-Conjugated Antibodies and Its Application in Multiplex Protein Detection in Single Cells”. 《Bioconjugate Chemistry》 27 (1): 217–25. January 2016. doi:10.1021/acs.bioconjchem.5b00613. PMID 26689321.
- ↑ 가 나 다 라 “Label-free Quantification of Proteins in Single Embryonic Cells with Neural Fate in the Cleavage-Stage Frog (Xenopus laevis) Embryo using Capillary Electrophoresis Electrospray Ionization High-Resolution Mass Spectrometry (CE-ESI-HRMS)”. 《Molecular & Cellular Proteomics》 15 (8): 2756–68. August 2016. doi:10.1074/mcp.M115.057760. PMC 4974349. PMID 27317400.
- ↑ 가 나 다 “Single Cell Proteomics Using Frog (Xenopus laevis) Blastomeres Isolated from Early Stage Embryos, Which Form a Geometric Progression in Protein Content”. 《Analytical Chemistry》 88 (13): 6653–7. July 2016. doi:10.1021/acs.analchem.6b01921. PMC 4940028. PMID 27314579.
- ↑ 가 나 다 라 “Identification of Maturation-Specific Proteins by Single-Cell Proteomics of Human Oocytes”. 《Molecular & Cellular Proteomics》 15 (8): 2616–27. August 2016. doi:10.1074/mcp.M115.056887. PMC 4974340. PMID 27215607.
- ↑ “SCoPE-MS -- We can finally do single cell proteomics!!!”. 《News in Proteomics Research》 (미국 영어). 2017년 3월 9일. 2017년 6월 28일에 확인함.
- ↑ “Single-cell proteomics – Slavov Lab Blog”. 《Slavov Lab Blog》 (미국 영어). 2017년 6월 6일. 2017년 6월 27일에 확인함.
- ↑ “Automated sample preparation for high-throughput single-cell proteomics”. 《bioRxiv》 (영어): 399774. 2018년 8월 25일. doi:10.1101/399774.
- ↑ “High-density micro-arrays for mass spectrometry”. 《Lab on a Chip》 10 (23): 3206–9. December 2010. doi:10.1039/C0LC00211A. PMID 20938499.
- ↑ “Universal sample preparation method for proteome analysis”. 《Nature Methods》 6 (5): 359–62. May 2009. doi:10.1038/nmeth.1322. PMID 19377485.
- ↑ “Global absolute quantification reveals tight regulation of protein expression in single Xenopus eggs”. 《Nucleic Acids Research》 42 (15): 9880–91. September 2014. doi:10.1093/nar/gku661. PMC 4150773. PMID 25056316.
- ↑ 가 나 다 “Single-cell metabolomics: analytical and biological perspectives”. 《Science》 342 (6163): 1243259. December 2013. doi:10.1126/science.1243259. PMID 24311695.
- ↑ “In situ metabolic analysis of single plant cells by capillary microsampling and electrospray ionization mass spectrometry with ion mobility separation”. 《The Analyst》 139 (20): 5079–85. October 2014. Bibcode:2014Ana...139.5079Z. doi:10.1039/C4AN01018C. PMID 25109271.[깨진 링크(과거 내용 찾기)]
- ↑ “Advances in mass spectrometry based single-cell metabolomics”. 《The Analyst》 144 (3): 782–793. January 2019. Bibcode:2019Ana...144..782D. doi:10.1039/C8AN01581C. PMID 30426983.
참고 문헌 편집
- Lim, S. B., Lim, C. T., & Lim, W. T. (2019). Single-cell analysis of circulating tumor cells: why heterogeneity matters. Cancers, 11(10), 1595. PMID 31635038PMID 31635038 PMC 6826423 doi 10.3390/cancers11101595
- Zhou, W. M., Yan, Y. Y., Guo, Q. R., Ji, H., Wang, H., Xu, T. T., ... & Zhang, J. Y. (2021). Microfluidics applications for high-throughput single cell sequencing. Journal of Nanobiotechnology, 19(1), 1-21. PMID 34635104PMID 34635104 PMC 8507141 doi 10.1186/s12951-021-01045-6
- Ding, L., Radfar, P., Rezaei, M., & Warkiani, M. E. (2021). An easy-to-operate method for single-cell isolation and retrieval using a microfluidic static droplet array. Microchimica Acta, 188(8), 242. PMID 34226955PMID 34226955 doi 10.1007/s00604-021-04897-9
- Luo, C., Liu, H., Xie, F., Armand, E. J., Siletti, K., Bakken, T. E., ... & Ecker, J. R. (2022). Single nucleus multi-omics identifies human cortical cell regulatory genome diversity. Cell Genomics, 2(3), 100107. https://doi.org/10.1016/j.xgen.2022.100107 Single-cell multi-omic method, simultaneously profiling transcriptome, DNA methylome, and chromatin accessibility
- Descamps, L., Le Roy, D., & Deman, A. L. (2022). Microfluidic-Based Technologies for CTC Isolation: A Review of 10 Years of Intense Efforts towards Liquid Biopsy. International Journal of Molecular Sciences, 23(4), 1981. PMID 35216097PMID 35216097 PMC 8875744 doi 10.3390/ijms23041981