분자유전학
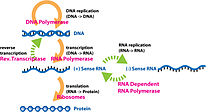
분자유전학(分子遺傳學, 영어: molecular genetics)은 유전학적 검사법을 사용하여 생명체의 게놈에서 유전자의 구조 또는 기능을 조사하는 유전학의 하위 분야이다.[1] 연구 분야는 고전적인 멘델의 유전법칙, 세포생물학, 분자생물학, 생화학 및 생명 공학 기술과 같은 생물학의 여러 하위 분야의 통합을 기반으로 한다. 연구자들은 유전자에서 돌연변이를 조사하거나 유전자에서 돌연변이를 유도하여 유전자 서열을 특정 표현형에 연관시킨다. 분자유전학을 통해 돌연변이를 유전 조건과 연관시켜 다양한 유전 질환에 대한 치료에 도움이 될 수 있다.
| 분자유전학 | |
|---|---|
 | |
| 학문명 | 분자유전학 |
역사 편집
분자유전학이 학문으로 발전하기 위해서는 몇 가지 과학적 발견이 필요했다. 유전 암호를 한 세포에서 다른 세포로, 그리고 세대 간에 전이시키는 수단으로서 DNA와 같은 유전을 담당하는 분자를 확인하는 것이 필수적이었다. 제임스 왓슨과 프랜시스 크릭은 로절린드 프랭클린과 모리스 윌킨스와 함께 분자유전학의 초석이라 할 수 있는 DNA의 구조를 알아냈다.[2] 1969년, 알버와 린이 대장균(E. coli)에서 제한 효소를 분리함으로써 유전공학 분야가 열렸다.[3] 제한 효소는 전기영동에 의한 분리를 위해 DNA를 선형화하기 위해 사용되었으며, 서던 블랏은 Hybridization probe를 통해 특정 DNA 조각을 식별 할 수 있게 해주었다.[4][5] 1971년, 베르그는 제한 효소를 사용하여 첫 번째 재조합 DNA 분자와 첫 번째 재조합 DNA 플라스미드를 만들었다.[6] 1972년에 코헨과 보이어는 재조합 DNA 플라스미드를 대장균에 삽입하여 최초의 재조합 DNA 유기체를 만들어 분자 복제의 길을 열었다.[7] 1970년대 후반, 맥삼과 길버트, 프레더릭 생어는 DNA 시퀀싱 기술을 개발하였다. 이는 분자유전자 연구에 중추였으며 과학자들이 유전자형 서열을 표현형과 관련시키기 위해 유전자 검사를 수행했다.[8] 1985년에 Mullis가 고안한 Taq 중합 효소를 사용한 중합효소 연쇄 반응(PCR)을 통해 특정 DNA 서열의 복사본을 만들 수 있었다.[9] 10년 후, 최초의 전체 게놈이 시퀀싱되었고(Haemophilus influenzae), 2001년 인간 게놈 프로젝트를 통해 인간 게놈의 최종 시퀀싱이 완성이 되었다.[10][11] 오늘날, 분자 유전 기술의 적용을 통해, 많은 모델 유기체에서 유전체학이 연구되고 있으며 NCBI 및 Ensembl과 같은 컴퓨터 데이터베이스에서 데이터가 수집되고 있다. 같은 종 내에서 그리고 다른 종들 사이의 유전자의 컴퓨터 분석 및 비교를 생물정보학(Bioinformatics)이라고 한다.
분자 유전학의 기술 편집
정유전학 편집
정유전학(Forward Genetics)은 특정 표현형을 생성하는 유전자 또는 유전자 돌연변이를 식별하는 데 사용되는 분자유전학 기술이다. 유전자적 검사법에서 돌연변이(화학 물질 또는 방사선) 또는 트랜스포손으로 무작위 돌연변이가 생성되고 개체는 특정 표현형에 대해 스크리닝된다. 원하는 표현형을 갖는 돌연변이 체가 비돌연변이 체로부터 선택되도록 항생제 저항성 유전자 또는 형광 리포터용 유전자를 사용하여 세포를 형질 전환 시킬 수 있다. 관심 표현형을 나타내는 돌연변이체를 분리하고, 표현형이 하나 이상의 유전자로부터 유래 하는지를 결정하기 위해 상보적 시험을 수행할 수 있다. 돌연변이 유전자는 우성(기능의 이득을 초래 함), 열성(기능의 상실을 나타냄) 또는 상위성(돌연변이 유전자가 다른 유전자의 표현형을 가리는 것)으로 특징지어진다. 마지막으로, 돌연변이의 위치와 특이성은 시퀀싱을 통해 매핑된다[12] 종종 정유전학은 예상치 못한 많은 발견으로 이어진다는 장점이 있지만, 많은 비용과 시간이 소요될 수 있다는 단점도 존재한다. 예쁜꼬마선충(Caenorhabditis elegans), 초파리(Drosophila melanogaster) 및 제브라피시(Danio rerio)와 같은 모델 유기체는 유전자 돌연변이로 인한 표현형을 연구하는데 사용된다.[13]
역유전학 편집
역유전학 (Reverse genetics)은 관심있는 유전자에서 의도적인 돌연변이로 인한 표현형을 결정하는 데 사용되는 분자유전학 기술이다. 표현형은 돌연변이 되지 않은 유전자의 기능을 추론하는 데 사용된다. 돌연변이는 관심 유전자에 대한 무작위적이거나 의도적인 변화 일 수 있다. 돌연변이는 뉴클레오타이드 치환, 프레임 시프트 돌연변이를 유도하기위한 뉴클레오타이드 첨가, 결실, 또는 유전자의 완전한 첨가/삭제에 의해 야기되는 돌연변이 일 수 있다. 특정 유전자의 결실은 유전자가 발현되지 않고 기능 결과 상실(예 : 녹아웃 마우스)의 유전자 제거(Gene Knock-Out)를 생성한다. 잘못 감지된 돌연변이는 기능의 전체 손실을 유발하거나 기능 저하의 일부를 잃을 수 있다. 녹다운은 또한 RNA 간섭(RNAi)에 의해 달성 될 수 있다.[15] 대안적으로, 유전자는 유기 유전자(트랜스진으로도 알려짐)로 치환되어 유전자 녹인(Gene Knock-In)을 생성하고 숙주에 의해 기능이 향상 될 수있다.[16] 비록 이들 기술이 표현형을 특정 기능에 연결시키는 결정에 관한 편견이 있지만, 관심 유전자가 이미 알려져 있기 때문에 정유전학보다 생산적인 측면에서는 훨씬 빠르다.
참고 편집
각주 편집
- ↑ Alberts, Bruce (2014년 11월 18일). 《Molecular biology of the cell》 Six판. New York, NY. ISBN 9780815344322. OCLC 887605755.
- ↑ Tobin, Martin J. (2003년 4월 15일). “April 25, 1953”. 《American Journal of Respiratory and Critical Care Medicine》 167 (8): 1047–1049. doi:10.1164/rccm.2302011. ISSN 1073-449X. PMID 12684243.
- ↑ “Restriction Enzymes Spotlight | Learn Science at Scitable”. 《www.nature.com》. 2019년 10월 7일에 확인함.
- ↑ Righetti, Pier Giorgio (2005년 6월 24일). “Electrophoresis: The march of pennies, the march of dimes”. 《Journal of Chromatography A》 1079 (1–2): 24–40. doi:10.1016/j.chroma.2005.01.018.
- ↑ “Southern Blotting | MyBioSource Learning Center”. 2019년 11월 11일에 원본 문서에서 보존된 문서. 2019년 11월 11일에 확인함.
- ↑ “Professor Paul Berg | Biographical summary”. 《WhatisBiotechnology.org》. 2019년 6월 19일에 원본 문서에서 보존된 문서. 2019년 10월 7일에 확인함.
- ↑ “Herbert W. Boyer and Stanley N. Cohen”. 《Science History Institute》. 2016년 6월 1일. 2019년 10월 7일에 원본 문서에서 보존된 문서. 2019년 10월 7일에 확인함.
- ↑ “DNA sequencing | genetics”. 《Encyclopedia Britannica》. 2019년 10월 7일에 확인함.
- ↑ “The Invention of PCR”. 《Bitesize Bio》. 2007년 10월 24일. 2019년 10월 7일에 원본 문서에서 보존된 문서. 2019년 10월 7일에 확인함.
- ↑ “Timeline: Organisms that have had their genomes sequenced”. 《yourgenome》. 2019년 10월 7일에 원본 문서에서 보존된 문서. 2019년 10월 7일에 확인함.
- ↑ “What is genomics?”. 《EMBL-EBI Train online》. 2011년 9월 9일. 2019년 10월 7일에 원본 문서에서 보존된 문서. 2019년 10월 7일에 확인함.
- ↑ Schneeberger, Korbinian (2014년 8월 20일). “Using next-generation sequencing to isolate mutant genes from forward genetic screens”. 《Nature Reviews Genetics》 (영어) 15 (10): 662–676. doi:10.1038/nrg3745. ISSN 1471-0056. PMID 25139187.
- ↑ Lawson, Nathan D.; Wolfe, Scot A. (2011년 7월 19일). “Forward and Reverse Genetic Approaches for the Analysis of Vertebrate Development in the Zebrafish”. 《Developmental Cell》 (영어) 21 (1): 48–64. doi:10.1016/j.devcel.2011.06.007. ISSN 1534-5807. PMID 21763608. 2013년 6월 24일에 원본 문서에서 보존된 문서. 2020년 1월 8일에 확인함.
- ↑ Kutscher, Lena M. (2014). “Forward and reverse mutagenesis in C. elegans”. 《WormBook》: 1–26. doi:10.1895/wormbook.1.167.1. PMC 4078664. PMID 24449699. 2019년 12월 3일에 원본 문서에서 보존된 문서. 2020년 1월 8일에 확인함.
- ↑ Hardy, Serge; Legagneux, Vincent; Audic, Yann; Paillard, Luc (October 2010). “Reverse genetics in eukaryotes”. 《Biology of the Cell》 102 (10): 561–580. doi:10.1042/BC20100038. PMC 3017359. PMID 20812916.
- ↑ Doyle, Alfred; McGarry, Michael P.; Lee, Nancy A.; Lee, James J. (April 2012). “The construction of transgenic and gene knockout/knockin mouse models of human disease”. 《Transgenic Research》 21 (2): 327–349. doi:10.1007/s11248-011-9537-3. ISSN 0962-8819. PMC 3516403. PMID 21800101.