핵산 분해 효소
핵산 분해 효소(核酸分解酵素, 영어: nuclease)는 핵산의 뉴클레오타이드 사이의 인산다이에스터 결합을 절단 할 수 있는 효소이다. 뉴클레이스, 핵산가수분해효소(核酸加水分解酵素)라고도 한다. 핵산 분해 효소는 표적 분자에서 단일 및 이중 가닥 파괴에 다양한 영향을 미친다. 살아있는 유기체에서 DNA 수선에 필수적인 요소이다. 특정 핵산 분해 효소의 결함은 유전적 불안정성, 면역 결핍을 유발할 수 있다.[1] 또한 핵산 분해 효소는 분자 클로닝에 광범위하게 사용된다.[2]
효소의 활동 장소에 따라 두 가지 분류가 존재한다. 핵산 외부 가수분해 효소(Exonuclease)는 말단으로부터 핵산을 분해시킨다. 핵산 중간 분해 효소(Endonuclease)는 표적 분자의 중간 부분에서 작용한다. 그들은 DNA 분해 효소(Deoxyribonucleases)와 RNA 분해 효소(Ribonuclease)로 더 세분화된다. 전자는 DNA에, 후자는 RNA에 작용한다.[2]
역사
편집1960년대 후반, 과학자 스튜어트 린(Stuart Linn)과 베르너 아르버(Werner Arber)는 대장균(E. coli)에서 성장 제한을 담당하는 두 가지 유형의 효소를 분리했다.[3][4] 이들 효소 중 하나는 DNA에 메틸기를 첨가하여 메틸화 된 DNA를 생성하는 한편, 다른 하나는 분자 길이를 따라 다양한 위치에서 메틸화되지 않은 DNA를 절단하였다. 첫 번째 유형의 효소는 '메틸레이즈(Methylase)라고 하고 다른 하나는 '제한 뉴클리에이즈(Restriction Nuclease)'로 불렸다. 이 효소는 DNA 분자를 자르고 붙여넣기하는 데 필요한 효소를 모으는 과학자들에게 중요했다. 그 당시에는 과학자들이 분자 길이에 따라 임의의 부위가 아닌 특정 부위에서 DNA를 절단하여 과학자들이 예측 가능하고 재현 가능한 방식으로 DNA 분자를 절단 할 수 있는 도구가 필요했다.
1968년, 존스 홉킨스 대학교에서 일했던 HO Smith, KW Wilcox, TJ Kelley는 기능이 특정 DNA 뉴클레오타이드 서열에 의존하는 최초의 제한 뉴클리에이즈를 분리했다. 헤모필루스 인플루엔자를 사용하여 Hind II라고 불리는 효소를 분리하여 6개의 염기쌍의 특정 서열 내 특정 지점에서 DNA 분자를 절단했다. 그들은 Hind II효소가 항상이 서열의 중심에서 직접 절단한다는 것을 발견했다(3번째와 4번째 염기쌍 사이).
수치 분류 시스템
편집대부분의 핵산 분해 효소는 국제 생화학·분자생물학 연합 명칭위원회의 효소 번호에 의해 가수 분해 효소 (EC 3)로 분류된다. 핵산 분해 효소는 인산다이에스터 분해 효소(Phosphodiesterase), 라이페이스(Lipase) 및 인산 가수 분해 효소(Phosphatase)와 같은 가수 분해 효소의 서브 그룹인 에스터 가수 분해 효소(Esterase, EC 3.1)에 속한다. 핵산 분해 효소가 속하는 에스터 가수 분해 효소는 EC 3.1.11 - EC 3.1.31로 분류된다.
구조
편집핵산 분해 효소의 1차 구조는 활성 부위에서 크게 저조하게 보존되고 최소로 보존되며, 그 표면은 주로 산성 및 염기성 아미노산 잔기를 포함한다. 핵산 분해 효소는 접히는 단백질로 분류된다.[5]
장소 인식
편집핵산 분해 효소는 핵산과 연관있는 효소이다. 핵산 분해 효소는 그들의 인식 및 결합 방식에서 비특이적 및 특이적 결합을 다양하게 사용한다. 살아있는 유기체에서 두 가지 종류의 결합은 DNA 수선에서 중요한 역할을 한다.[6]
DNA 수선과 관련된 비특이적 핵산 중간 분해 효소는 표적 서열 또는 손상에 대해 DNA를 스캔 할 수 있다. 이러한 핵산 분해 효소는 표적과 마주 칠 때까지 DNA를 따라 확산되며, 활성 자리의 잔기가 DNA의 화학기와 상호 작용한다. EcoRV, BamHI, PvuII와 같은 핵산 중간 분해 효소의 경우, 이 비특이적 결합은 단백질의 최소 표면과 DNA 사이의 정전기적 상호 작용을 포함한다. 이 약한 결합은 DNA의 전체적인 형태가 변형되지 않은 상태로 남기 때문에 B형으로 남게된다.[6]
대조적으로 장소 특이적 핵산 분해 효소는 훨씬 더 강한 연관성을 형성한다. 이것은 DNA 결합 도메인의 깊은 홈으로 끌어들인다. 이는 DNA 3차 구조에 상당한 변형을 초래하고 염기성(양으로 하전 된) 잔기가 풍부한 표면으로 도달된다. DNA와의 광범위한 정전기 상호 작용에 관여한다.[6]
DNA 수선에 관여하는 일부 핵산 분해 효소는 부분 서열 특이성을 나타낸다. 그러나 대부분은 비특이적이며, 염기쌍 불일치에 의해 DNA 골격에서 생성된 구조적 이상을 인식한다.[6]
서열 특이적 핵산 분해 효소
편집| 효소 | 출처 | 인식 순서 | 절단 |
|---|---|---|---|
| HindII | Haemophilus influenzae | 5'–GTYRAC-3 ' </br> 3'–CARYTG–5 ' | 5'– GTY RAC –3 ' </br> 3'– CAR YTG –5 ' |
| R = A 또는 G ; Y = C 또는 T | |||
HindII의 초기 발견 이후, 230개 이상의 세균 균주로부터 일부 서열 특이적이거나 일부가 아닌 900개 이상의 제한 효소가 존재한다. 이러한 제한 효소는 일반적으로 자신의 기원을 반영하는 이름에서 온다. 예를 들어, <i id="mwjQ">Eco</i>RI는 대장균 RY13 박테리아에서 유래하고, HindII는 Haemophilus influenzae 균주 Rd에서 유래된다. 핵산 분해 효소 이름 뒤에 나오는 숫자는 효소가 박테리아의 단일 계통에서 분리 된 순서를 나타낸다(EcoRI, EcoRII).
핵산 중간 분해 효소
편집제한 핵산 중간 분해 효소(Restriction Endonulease)는 DNA 분자의 길이를 스캔함으로써 기능한다. 그것이 특정한 특이적 인식 서열을 만나면, DNA 분자에 결합하여 2개의 당-인산 골격에서 한 개의 절단을 만든다. 이들 절단의 위치는 서로 관련되어 있고, 인식 서열 자체와 제한 핵산 중간 분해 효소의 동일성에 의해 결정된다. 상이한 핵산 중간 분해 효소는 상이한 세트의 절단을 생성하지만, 하나의 핵산 중간 분해 효소는 작용하는 DNA 분자에 관계없이 항상 특정 염기 서열을 동일한 방식으로 절단한다. 절단이 완료되면 DNA 분자가 조각으로 나뉜다.
접착성 말단
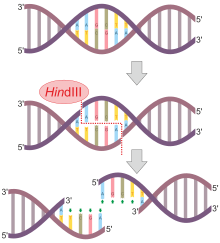
편집모든 제한 핵산 중간 분해 효소가 대칭적으로 절단되지 않고 위와 같이 기술된 HindII와 같이 무딘 말단(Blunt End)을 남기는 것은 아니다. 많은 중간 분해 효소는 DNA 골격을 서로 직접 대향하지 않는 위치에서 절단하여 돌출부를 생성한다. 예를 들어 핵산 분해 효소 EcoRI는 인식 서열 5'—GAATTC—3'을 들 수 있다.
| 효소 | 출처 | 인식 순서 | 절단 |
|---|---|---|---|
| HindIII | Haemophilus influenzae | </br> 3'–TTCGAA–5 ' |
</br> 3'– TTCGA A –5 ' |
| EcoRI | Escherichia coli | </br> 3'–CTTAAG–5 ' |
</br> 3 '– CTTAA G –5' |
| BamHI | Bacillus amyloliquefaciens | </br> 3'–CCTAGG–5 ' |
</br> 3'– CCTAG G –5 ' |
효소가 이 서열을 만나면, G와 가장 가까운 A 염기 잔기 사이의 각 골격을 절단한다. 절단이 이루어지면, 생성 된 단편은 상보적 염기를 서로 고정시키는 비교적 약한 수소 결합에 의해서만 함께 유지된다. 이들 결합의 약점은 DNA 단편이 서로 분리되도록 한다. 각각의 생성 된 단편은 짝을 이루지 않은 염기로 구성된 돌출 된 5' 말단을 갖는다. 다른 효소는 DNA 골격에서 절단을 생성하여 3' 말단이 튀어 나오게한다. 튀어 나온 끝(3' 말단 혹은 5' 말단)을 접착성 말단이라고 한다. 이어서 연결 효소(Ligase)를 사용하여 두 분자의 인산염 골격을 연결시킨다.
자연에서의 역할
편집DNA 수선
편집유전자 정보의 매개체로서 DNA에 의존하는 모든 세포에서, 유전자 품질 관리는 모든 유기체의 필수 기능이다. DNA 복제는 오류가 발생하기 쉬운 과정이며 DNA 분자 자체는 많은 신진 대사 및 환경 스트레스 요인에 의해 변형되기 쉽다. 일반적인 예는 활성산소 종, 근자외선, 이온화 방사선이 포함된다. 많은 핵산 분해 효소는 손상 부위를 인식하고, 이를 주변 DNA로부터 절단함으로써 DNA 수선에 참여한다. 이 효소는 각각 독립적으로 또는 복합체를 이루어 작동한다. DNA 수선에 관여하는 대부분의 핵산 분해 효소는 서열 특이적이지 않다. 이중 가닥 DNA(dsDNA) 2차 구조의 변형을 통해 손상 부위를 인식한다.[5]
복제 교정
편집DNA 복제 동안, DNA 중합효소는 상보적인 주형 가닥에 대해 새로운 DNA 가닥을 연장시킨다. 대부분의 DNA 중합 효소는 중합 효소 도메인과 교정 핵산 외부 가수분해 효소 도메인을 포함한다. 중합 효소는 새로운 가닥을 5' 말단 → 3' 말단 방향으로 연장시킨다. 핵산 외부 가수분해 효소는 3' 말단 → 5' 말단 방향으로 동일한 가닥으로부터 잘못된 뉴클레오타이드를 제거한다. 핵산 외부 가수분해 효소의 활성은 DNA 중합 효소의 교정 능력에 필수적이다. 이들 핵산 분해 효소를 불활성화 시키거나 제거하는 유전자 결실은 돌연변이 및 사망률을 증가시킨다.[7]
정지된 복제 기점
편집많은 형태의 DNA 손상으로 인해 복제 기점의 진행이 중단되어 DNA 중합 효소 및 관련 요소가 기점을 버린다. 그런 다음 기점 관련 단백질로 처리해야 한다. 가장 주목할만한 것은 MUS81이다. 감수성 결손 이외에 효모에서 UV 또는 메틸화 손상 민감성을 유발하는 결실이다.[5]
오카자키 절편 처리
편집세포에서 일반적인 작업으로 복제에서 오카자키 절편의 프라이머를 제거하는 것이다. 대부분의 이러한 프라이머는 RNase H의 핵산 중간 분해 효소에 의해 새로 합성 된 지연 가닥 DNA로부터 절제된다. 또한 진핵생물 및 고균에서, 플랩 핵산 분해 효소 FEN1은 오카자키 절편의 가공에 참여한다.[5]
불일치 수리
편집유기체에서 DNA 불일치 수선은 불일치 특이적 핵산 중간 분해 효소에 의해 영향을 받는다. 원핵 생물에서 이 역할은 주로 MutSLH 및 매우 짧은 패치 복구(VSP 복구) 관련 단백질에 의해 채워진다.
MutSLH 시스템(MutS, MutL 및 MutH 포함)은 점 돌연변이 및 작은 회전을 수정한다. MutS는 MutL 및 MutH를 모집하는 불일치를 인식하고 결합한다. MutL은 MutS와 MutH 사이의 상호 작용을 매개하고 후자의 핵산 중간 분해 효소의 활성을 향상시킨다. MutH는 반메틸화 된 5'—GATC—3' 부위를 인식하고 비메틸화 된 가닥(가장 최근에 합성 된 가닥)의 G옆에서 절단한다.
이중 가닥 파손 수리
편집의도적이든 비의도적이든 이중 가닥 손상은 세포에서 정기적으로 발생한다. 의도하지 않은 손상은 일반적으로 이온화 방사선, 다양한 외인성 및 내인성 화학 물질, 정지된 복제 기점에 의해 생성된다. 의도적인 손상은 감수 분열과 V(D)J 재조합의 중개자로서 생성되는데, 이는 주로 상동 재조합 과 비상동말단연결을 통해 복구된다. 두 경우 모두 수리가 이루어지기 전에 핵산 분해 효소로 이중 가닥 절단의 끝을 처리해야 한다. 하나의 이러한 핵산 분해 효소는 Rad50과 복합된 Mre11이다.
V(D)J 재조합은 이중 가닥 파괴와 관련된 스템-루프 구조를 개방하고 양단을 연결하는 것을 포함한다. Artemis-DNAPKcs복합체가 이 반응에 참여한다. 아르테미스는 단독으로 5' → 3' ssDNA 핵산 외부 가수분해 효소를 나타내지만, DNA-PKcs와의 복합체는 스템-루프 구조의 혈관 내 처리를 허용한다. 단백질 중 하나의 결함은 심각한 면역 결핍을 유발한다.[8]
한편 상동 재조합은 D-루프 또는 홀리데이 접합에 의해 연결된 2개의 상동 DNA 이중체를 포함한다. 세균에서 같은 핵산 중간 분해 효소 RuvC의 접합 센터 근처에 두 개의 대칭 장소에서 접합을 절단하여 두 개의 별도 dsDNAs로 해결한다. 진핵 생물에서 FEN1, XPF-ERCC1 및 MUS81 은 D-루프를 절단하고, Cce1/Ydc2는 미토콘드리아에서 홀리데이 접합을 처리한다.[8]
메가 핵산 분해 효소
편집특정 핵산 분해 효소가 주어진 DNA 분자를 절단하는 빈도는 DNA의 복잡성 및 핵산 분해 효소 인식 서열의 길이에 의존한다. 예를 들어, 주어진 4 염기 서열은 평균적으로 256 염기 쌍마다 (4^4 = 256) 발생할 것으로 예상되지만, 임의의 주어진 6 염기 서열이 예상 될 것이다 평균 4,096 개의 염기쌍마다 한 번씩 발생한다(4^6 = 4096).
핵산 분해 효소의 하나의 독특한 군은 메가 핵산 분해 효소이며, 이는 12~40개의 염기쌍으로 구성 된, 더 크고 덜 일반적인 인식 서열을 갖는 것을 특징으로 한다. 이 핵산 분해 효소는 식물 및 포유동물과 같은 복잡한 유기체에서의 유전자 공학 및 게놈 공학 응용에 특히 유용하며, 전형적으로 더 큰 게놈(수십억 개의 염기쌍의 수)은 전통적인 핵산 분해 효소를 사용하여 빈번하고 유해한 부위 특이적 분해를 야기시킬 수 있다.
같이 보기
편집- 핵산 분해 효소 방어 분석법
- 미구균 핵산 분해 효소
- S1 핵산 분해 효소
- P1 핵산 분해 효소
- HindIII
- PIN 도메인
각주
편집- ↑ Nishino, Tatsuya; Morikawa, Kosuke (2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. 《Oncogene》 21 (58): 9022–9032. doi:10.1038/sj.onc.1206135. PMID 12483517.
- ↑ 가 나 Rittié, Laure; Perbal, Bernard (2008). “Enzymes used in molecular biology: a useful guide”. 《Journal of Cell Communication and Signaling》 2 (1): 25–45. doi:10.1007/s12079-008-0026-2. PMC 2570007. PMID 18766469.
- ↑ Linn S., Arber, W. (1968). Host specificity of DNA produced by Escherichia coli, X. In vitro restriction of phage fd replicative form. Proc. Natl. Acad. Sci. USA. 59:1300-1306
- ↑ Arber, W., Linn S. (1969) DNA modification and restriction. Annu. Rev. Biochem. 38:467-500
- ↑ 가 나 다 라 Nishino, Tatsuya; Morikawa, Kosuke (2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. 《Oncogene》 21 (58): 9022–32. doi:10.1038/sj.onc.1206135. PMID 12483517.
- ↑ 가 나 다 라 Nishino, Tatsuya; Morikawa, Kosuke (2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. 《Oncogene》 21 (58): 9027–9028. doi:10.1038/sj.onc.1206135. PMID 12483517.
- ↑ Nishino, Tatsuya; Morikawa, Kosuke (2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. 《Oncogene》 21 (58): 9022, 9023. doi:10.1038/sj.onc.1206135. PMID 12483517.
- ↑ 가 나 Nishino, Tatsuya; Morikawa, Kosuke (2002). “Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors”. 《Oncogene》 21 (58): 9024, 9025. doi:10.1038/sj.onc.1206135. PMID 12483517.
외부 링크
편집- 위키미디어 공용에 핵산 분해 효소 관련 미디어 분류가 있습니다.