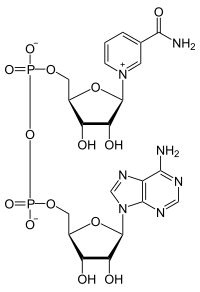
니코틴아마이드 아데닌 다이뉴클레오타이드
니코틴아마이드 아데닌 다이뉴클레오타이드(영어: nicotinamide adenine dinucleotide, NAD)는 모든 살아있는 세포에서 발견되는 보조 인자이다. NAD는 인산기를 통해 결합된 두 개의 뉴클레오타이드로 구성되어 있기 때문에 다이뉴클레오타이드라고 불린다. 뉴클레오타이드 중 하나는 아데닌 핵염기를 가지고, 다른 하나는 니코틴아마이드를 가지고 있다. NAD는 산화형과 환원형의 두 가지 형태로 존재하며, 산화형은 NAD+, 환원형은 NADH로 약칭된다.
| |

| |
| 이름 | |
|---|---|
| 별칭
diphosphopyridine nucleotide (DPN틀:+),
coenzyme I | |
| 식별자 | |
3D 모델 (JSmol)
|
|
| ChEBI | |
| ChEMBL | |
| ChemSpider | |
| DrugBank | |
| ECHA InfoCard | 100.000.169 |
| KEGG | |
PubChem CID
|
|
| RTECS 번호 |
|
| UNII |
|
| |
| |
| 성질 | |
| C21H27N7O14P2 | |
| 몰 질량 | 663.43 g/mol |
| 겉보기 | 흰색 분말 |
| 녹는점 | 160 °C (320 °F; 433 K) |
| 위험 | |
| 주요 위험 | 위험하지 않음 |
| NFPA 704 (파이어 다이아몬드) | |
달리 명시된 경우를 제외하면, 표준상태(25 °C [77 °F], 100 kPa)에서 물질의 정보가 제공됨.
| |
물질대사에서 NAD는 산화환원 반응에 관여하며, 한 반응에서 다른 반응으로 전자를 전달한다. 따라서 NAD는 세포에서 두 가지 형태로 발견된다. NAD+는 산화제로서 다른 분자로부터 전자를 받아서 환원된다. 환원된 NAD+는 NADH가 되어 전자를 공여할 수 있는 환원제로 사용될 수 있다. 이러한 전자전달 반응은 NAD의 주요 기능이다. 또한 이것은 다른 세포 과정에서도 사용되며, 특히 번역 후 변형에서 단백질로부터 작용기를 추가하거나 제거하는 효소들의 기질에 사용된다. 이러한 기능의 중요성 때문에 NAD 대사에 관여하는 효소들은 신약 개발의 표적이 된다.
생물체에서 NAD는 아미노산인 트립토판이나 아스파르트산으로부터 합성될 수 있다. 다른 방식으로 조효소의 보다 복잡한 구성 성분들이 니아신처럼 음식물로부터 섭취된다. 유사한 화합물들이 NAD의 구조를 파괴하는 반응에 의해 방출된다. 그런 다음 이들 예비 화합물들은 회수 경로(salvage pathway)를 거치면서 활성형으로 재활용된다. 일부 NAD는 니코틴아마이드 아데닌 다이뉴클레오타이드 인산(NADP)으로 전환된다. NADP는 NAD와 비슷하지만, 물질대사에서 다른 역할을 수행한다.
NAD+는 특정 질소 원자에 대한 형식 전하 때문에 위첨자 "+"로 표기되었지만, 생리학적 pH에서 대부분은 1가 음이온이고, NADH는 2개의 연결된 인산기 때문에 2가 음이온이다.
물리적 특성 및 화학적 특성
편집모든 다이뉴클레오타이드와 마찬가지로 NAD도 2개의 연결된 인산기에 의해 결합된 2개의 뉴클레오사이드로 구성된다. 각각의 뉴클레오사이드는 리보스의 1' 위치에 아데닌이 결합되어 있고, 다른 하나는 니코틴아마이드가 결합되어 있다. 니코틴아마이드 잔기는 리보스의 아노머 탄소에 2가지 방향으로 부착될 수 있다. 이러한 2가지 가능한 구조 때문에 화합물은 2가지 부분입체 이성질체로 존재한다. 생물체에서 발견되는 것은 NAD+의 β-니코틴아마이드 부분입체 이성질체이다. 이들 뉴클레오타이드들은 5' 탄소를 통해 2개의 인산기 가교로 서로 결합된다.[1]
물질대사에서 NAD는 산화환원반응에서 전자를 공여하거나 수용한다.[2] 이러한 반응(아래의 화학식에서 요약됨)은 하이드라이드(H−) 및 양성자(H+)의 형태로 반응물로부터 2개의 수소 원자를 제거하는 것을 포함한다. 양성자는 용액으로 방출되는 반면 환원제 RH2는 산화되고, NAD+는 니코틴아마이드 고리로의 하이드라이드의 전달에 의해 NADH로 환원된다.
- RH2 + NAD+ → NADH + H+ + R
하이드라이드 전자쌍으로부터 1개의 전자는 NAD+의 니코틴아마이드 고리의 양(+)으로 하전된 질소로 전달되고, 두 번째 수소 원자는 이 질소의 반대편의 C4 탄소 원자로 전달된다. NAD+/NADH 산화환원쌍의 표준환원전위는 -0.32 볼트이며, 이는 NADH를 강력한 환원제로 만든다.[3] NADH가 다른 분자를 환원시키고, NAD+로 재산화되기 때문에 반응은 가역적이다. 이것은 조효소가 소모되지 않고 NAD+와 NADH 형태 사이를 계속적으로 순환할 수 있다는 것을 의미한다.[1]
외견상, NAD의 모든 형태는 흡습성이 있고, 물에 잘 녹는 흰색의 무정형 분말이다.[4] NAD는 건조하고 어두운 곳에 보관하면 안정적이다. NAD+ 용액은 4 °C 및 중성 pH에서 약 1주일 동안 무색이며 안정적이지만, 산이나 알칼리에서는 빠르게 분해된다. NAD는 분해시 효소 저해제인 생성물을 형성한다.[5]
뉴클레오타이드가 기본 골격이기 때문에 흡수 피크가 260 나노미터로 DNA와 같다. 340 nm의 파장은 NADH만이 잘 흡수하기 때문에 NAD+ ⇄ NADH의 변화(=탈수소효소의 활성도 측정)는 340 nm에서의 흡광도 측정에 의하여 조사할 수 있다. NAD+와 NADH는 모두 아데닌 때문에 자외선을 잘 흡수한다. 예를 들어 NAD+의 흡수 피크는 260 nm의 파장에 있고, 흡광계수는 16,900 M−1cm−1이다. NADH는 더 긴 파장인 340 nm에서 두 번째 자외선 흡수 피크를 나타내고, 흡광계수는 6,220 M−1cm−1이다.[6] 더 긴 파장에서 조효소의 산화된 형태와 환원된 형태 사이의 자외선 흡수 스펙트럼의 차이는 분광광도계를 사용하여 340 nm에서 자외선 흡수량을 측정하여 효소 분석에서 서로 간의 전환율을 간단히 측정할 수 있도록 한다.[6]
또한 NAD+와 NADH는 형광에서도 다르다. 용액 속의 NADH는 460 nm에서 발광 피크 및 0.4 나노초의 형광 수명을 가지는 반면에 NAD+는 형광을 나타내지 않는다.[7] NADH가 단백질과 결합할 때 형광 신호의 특성은 변하기 때문에, 이러한 변화는 효소반응속도론을 연구하는데 유용한 해리상수를 측정하는데 사용될 수 있다.[7][8] 이러한 형광의 변화는 형광 현미경을 통해 살아있는 세포의 산화환원 상태 변화를 측정하는 데에도 사용된다.[9]
세포 내 농도 및 상태
편집쥐의 간에서 NAD+와 NADH의 총량은 습윤 중량의 그램 당 약 1 μ몰이며, 동일한 세포에서 NADP+와 NADPH의 농도는 약 10배이다.[10] 세포기질에서 NAD+의 실제 농도는 측정하기가 더 힘든데, 동물세포에서 최근의 추정치는 약 0.3mM이고,[11][12] 효모에서는 1.0~2.0 mM이다.[13] 그러나 미토콘드리아에서 NADH 형광의 80% 이상이 결합된 형태여서 용액 중의 농도는 훨씬 낮다.[14]
미토콘드리아에서 NAD+의 농도는 세포기질의 농도와 비슷하지만, 세포 내 다른 구획들에 대한 데이터는 제한적이다.[12] 이 NAD+는 조효소가 세포막을 가로질러 확산될 수 없기 때문에 특정 세포막 운반 단백질에 의해 미토콘드리아로 운반된다.[15]
NAD의 산화형(NAD+)과 환원형(NADH) 사이의 균형을 NAD+/NADH 비율이라고 한다. 이 비율은 세포의 산화환원 상태와 세포의 대사 홀성과 건강을 반영하는 중요한 요소이다.[16] NAD+/NADH 비율의 영향은 복잡하며, 글리세르알데하이드 3-인산 탈수소효소 및 피루브산 탈수소효소를 비롯한 여러 주요 효소들의 활성을 조절한다. 건강한 포유류의 조직에서 세포질의 유리 NAD+와 NADH 사이의 비율은 일반적으로 약 700이며, 이 비율은 산화반응에 유리하다.[17][18] 전체 NAD+/NADH의 비율은 훨씬 더 낮으며, 포유류의 경우 3~10 에 이를 것으로 추정된다.[19] 대조적으로 NADP+/NADPH 비율은 일반적으로 약 0.005이므로, NADPH가 보다 더 지배적인 형태이다.[20] 이러한 다른 비율은 NADH와 NADPH의 서로 다른 대사상의 역할에 핵심이다.
생합성
편집NAD+는 두 가지 대사 경로를 통해 합성된다. NAD+는 아미노산과 같은 간단한 전구물질로부터 새롭게 생합성하는 데노보(de novo) 경로 또는 기존의 니코틴아마이드와 같은 구성 성분들을 재활용하여 NAD+를 합성하는 회수(salvage) 경로를 통해 합성된다.
데노보 경로
편집대부분의 생물체들은 단순한 구성 성분으로부터 NAD+를 합성한다.[2] 특정 반응들은 생물체마다 다르지만 공통적인 특징은 동물과 일부 세균에서 트립토판, 식물과 일부 세균에서 아스파르트산과 같은 아미노산으로부터 퀴놀린산을 생성하는 것이다.[21][22] 포스포리보스 잔기의 전달에 의해 퀴놀린산이 니코틴산 모노뉴클레오타이드(NaMN)로 전환된다. 이어서 아데닐산 잔기가 전달되어 니코틴산 아데닌 다이뉴클레오타이드(NaAD)를 형성한다. 마지막으로 니코틴산 아데닌 다이뉴클레오타이드의 니코틴산 부분은 니코틴아마이드로 아마이드화되어, NAD를 형성한다.[2]
추가적인 단계에서 일부 NAD+는 NAD+를 인산화시키는 NAD+ 키네이스에 의해 NADP+로 전환된다.[23] 결핵균(Mycobacterium tuberculosis)과 호열성 고균인 Pyrococcus horikoshii 와 같은 몇몇 세균들이 무기 폴리인산을 NAD+ 키네이스에 사용되는 인산기의 공급원으로 사용하지만, 대부분의 생물체들에서 NAD+ 키네이스는 ATP를 인산기의 공급원으로 사용한다.[24][25]
회수 경로
편집데노보 경로를 통해 간단한 아미노산 전구체로부터 NAD+를 합성하는 것 외에도 세포는 회수 경로를 통해 피리미딘 염기를 함유하는 화합물을 형성한다. 이러한 회수 경로에서 사용되는 3가지 비타민 전구물질들은 니코틴산(NA), 니코틴아마이드(Nam), 니코틴아마이드 리보사이드(NR)이다.[2] 이러한 화합물은 음식물을 통해 섭취할 수 있으며, 비타민 B3 또는 니아신이라고 불린다. 그러나 이들 화합물은 세포 내에서도 생성되고, 세포 내의 NAD+의 소화에 의해서도 생성된다. 회수 경로에 관여하는 효소들의 일부는 세포핵에 집중되어 있는 것처럼 보이는데 이는 세포핵에서 NAD+를 소비하는 높은 수준의 반응을 보상할 수 있다.[26] 포유류의 세포가 주변으로부터 세포 외 NAD+를 흡수할 수 있다는 보고가 있으며,[27] 니코틴아마이드와 니코틴아마이드 리보사이드는 둘 다 내장에서 흡수될 수 있다.[28]
데노보 경로가 존재함에도 불구하고 회수 경로는 사람에게 필수적이다. 식사에 니아신이 부족하면 펠라그라 같은 비타민 결핍증이다.[29] 산화환원반응에서 산화형과 환원형 사이의 NAD+의 순환이 조효소의 전체 수준을 변화시키지 않기 때문에, 번역 후 변형과 같은 반응에서 조효소의 지속적인 소비로 인해 높은 수준의 NAD+를 요구하게 된다.[2]
미생물에서 사용되는 회수 경로는 포유류의 회수 경로와 다르다.[30] 효모 Candida glabrata 와 인플루엔자균(Haemophilus influenzae)과 같은 일부 병원체들은 NAD+ 영양요구주들은 NAD+를 합성할 수는 없지만 회수 경로를 가지고 있기 때문에 NAD+ 또는 NAD+ 전구물질의 외부 공급원에 의존한다.[31][32] 더욱 놀라운 것은 세포 내 병원체인 클라미디아 트라코마티스(Chlamydia trachomatis)인데, 클라미디아 트라코마티스는 NAD+와 NADP+의 생합성이나 회수에 관련된 유전자를 인식할 수 있는 후보물질이 부족하고, 숙주로부터 NAD+와 NADP+를 확보해야 한다.[33]
기능
편집NAD는 물질대사에서 몇 가지 중요한 역할을 한다. NAD는 단백질로부터 아세틸기를 제거하기 위해 NAD+를 사용하는 시르투인이라는 효소와 세균의 DNA 리게이스의 기질 역할을 할 뿐만 아니라 2차 신호전달물질인 사이클릭 ADP-리보스(cyclic ADP-ribose)의 전구물질, ADP-리보실화(ADP-ribosylation) 반응에서 ADP-리보스(ADP-ribose) 잔기의 공여체로 산화환원반응의 조효소로 작용한다. 이러한 대사 기능 외에도 NAD+는 자발적으로 그리고 조절된 메커니즘에 의해 세포로부터 방출될 수 있는 아데닌 뉴클레오타이드로 나타나며,[35][36] 따라서 세포 외에서 중요한 역할을 할 수 있다.[36]
NAD와 산화환원효소의 결합
편집물질대사에서 NAD+의 주요 역할은 한 분자에서 다른 분자로 전자를 전달하는 것이다. 이 유형의 반응은 산화환원효소라고 불리는 효소 부류에 의해 촉매된다. 이러한 효소들의 정확한 명칭은 두 기질의 이름을 모두 포함한다. 예를 들어 NADH-유비퀴논 산화환원효소는 조효소 Q에 의해 NADH의 산화를 촉매한다.[37] 이러한 효소들은 탈수소효소 또는 환원효소라고 하며, NADH-유비퀴논 산화환원효소는 일반적으로 NADH 탈수소효소 또는 조효소 Q 환원효소로 불린다.[38]
NAD+/NADH에 결합하는 다양한 효소 부류들이 있다. 가장 일반적인 효소 부류들 중 하나는 로스만 폴드(Rossmann fold)로 알려진 구조적 모티프를 포함한다.[39][40] 이 모티프는 뉴클레오타이드 결합 단백질 내에서 이러한 구조가 얼마나 흔한지를 처음으로 발견한 과학자인 마이클 로스만(Michael Rossmann)의 이름을 따서 지어졌다.[41]
그러나 세균에서 아미노산 대사에 관여하는 NAD-결합 효소는 로스만 폴드를 가지고 있지 않다.[42]
산화환원효소의 활성 부위에 결합할 때, NAD의 니코틴아마이드 고리는 다른 기질로부터 하이드라이드를 수용할 수 있도록 위치하게 된다. 효소에 따라 하이드라이드 공여체는 그림에서 나타난 것처럼 C4 탄소 평면의 위쪽 또는 아래쪽에 위치한다. 클래스 A 산화환원효소는 위쪽에서 원자를 이동시키고, 클래스 B 산화환원효소는 아래쪽에서 원자를 이동시킨다. 수소를 받아들이는 C4 탄소가 프로키랄(prochiral)이기 때문에, 효소에 대한 메커니즘에 대한 정보를 얻기 위해 효소반응속도론에서 이를 이용할 수 있다. 이는 수소를 대체하는 중수소 원자를 갖는 기질과 효소를 혼합함으로써 이루어지기 때문에, 효소는 수소보다 중수소를 전달해서 NAD+를 환원시킬 것이다. 이런 경우에 효소는 NADH의 두 개의 입체 이성질체 중 하나를 생성할 수 있다.[43]
단백질이 두 조효소와 결합하는 방식의 유사함에도 불구하고, 효소는 거의 항상 NAD+ 또는 NADP+에 대해 높은 수준의 특이성을 보여준다.[44] 이러한 특이성은 각각의 조효소들의 구별되는 대사적 역할을 반영하며, 2가지 유형의 조효소-결합 포켓(coenzyme-binding pocket) 내의 아미노산 잔기의 차이로 나타난다. 예를 들어 NADP-의존성 효소들의 활성 부위에서 염기성 아미노산 곁사슬과 NADP+의 산성 인산기 사이에 이온 결합이 형성된다. 반대로 NAD-의존성 효소들에서는 포켓의 전하가 역전되어, NADP+가 결합하는 것을 방지한다. 그러나 이 일반적인 규칙에는 몇 가지 예외가 있으며, 일부 생물 종에서 알도스 환원효소, 포도당 6-인산 탈수소효소 및 메틸렌테트라하이드로폴산 환원효소와 같은 효소는 NAD와 NADP 둘 다를 사용할 수 있다.[45]
산화환원반응에서의 역할
편집산화환원효소에 의해 촉매되는 산화환원반응은 물질대사의 모든 부분에서 필수적이지만, 이러한 반응이 일어나는 특히 중요한 부분 중 하나는 영양소로부터 에너지를 방출하는 것이다. 여기서 포도당과 지방산과 같은 환원된 화합물이 산화되어 에너지를 방출한다. 이 에너지는 베타 산화, 해당과정, 시트르산 회로를 통해 NAD+로 전달되어 NADH로 환원된다. 진핵생물의 세포질에서 생성된 NADH에 의해 운반되는 전자는 말산-아스파르트산 셔틀과 같은 미토콘드리아의 셔틀에 의해 미토콘드리아 내로 전달된다.[46] 미토콘드리아의 NADH는 전자전달계에 의해 차례로 산화되는데, 전자전달계는 미토콘드리아 내막을 가로질러 양성자를 미토콘드리아 기질로부터 막 사이 공간으로 펌핑하고, 산화적 인산화를 통해 ATP를 생성한다.[47] 이러한 셔틀 시스템은 또한 엽록체에서 동일한 수송 기능을 가지고 있다.[48]
NAD의 산화형(NAD+)과 환원형(NADH) 둘 다가 이러한 일련의 반응들에서 사용되기 때문에, 세포는 NAD+와 NADH 둘 다를 상당한 농도로 유지하며, NAD가 산화제와 환원제의 역할을 모두 수행할 수 있도록 높은 NAD+/NADH 비율을 유지한다.[49] 대조적으로 NADPH의 주된 기능은 지방산의 합성, 광합성과 같은 동화작용에서 환원제로 사용되는 것이다. 강력한 환원제로서 산화환원반응을 일으키기 위해서는 NADPH가 필요하기 때문에, NADP+/NADPH 비율은 매우 낮게 유지된다.[49]
NADH는 이화작용에서 중요하지만, NADH는 또한 포도당신생합성과 같은 동화작용에서도 사용된다.[50] 동화작용에서 NADH에 대한 필요성은 소량의 에너지만을 방출하는 영양소 위에서 자라는 원핵생물에게 문제를 야기한다. 예를 들어, 니크로박터(Nitrobacter)와 같은 질화 세균은 아질산염을 질산염으로 산화시켜 양성자를 펌핑하고, ATP를 생성하기에 충분한 에너지를 방출하지만, NADH를 직접 생성하기에는 에너지가 충분하지 않다.[51] NADH는 동화작용에 필요하기 때문에 이들 세균은 전자전달계의 일부를 역방향으로 작동시켜 NADH를 생성할 수 있는 충분한 양성자 구동력(proton-motive force)을 생성하기 위해 아질산염 산화환원효소를 사용한다.[52]
비산화환원반응에서의 역할
편집조효소인 NAD+는 ADP-리보스 전이반응에서도 소모된다. 예를 들어 ADP-리보실트랜스퍼레이스라고 불리는 효소는 ADP-리보실화라고 불리는 번역 후 변형에서 NAD+의 ADP-리보스 잔기를 단백질에 첨가한다.[53] ADP-리보실화는 모노 ADP-리보실화에서 단일 ADP-리보스 잔기의 첨가 또는 폴리 ADP-리보실화라고 불리는 긴 분지형 사슬을 만드는 과정에서 단백질로 ADP-리보스가 전이되는 것을 포함한다.[54] 모노 ADP-리보실화는 세균의 독소들, 특히 콜레라 독소의 메커니즘으로 처음 확인되었지만, 정상적인 세포 신호전달에도 관여한다.[55][56] 폴리 ADP-리보실화는 폴리 ADP-리보스 중합효소에 의해 수행된다.[54][57] 폴리 ADP-리보스 구조는 세포에서 일어나는 몇 가지 일들을 조절하는데 관여하며, 세포핵에서 일어나는 DNA 복구 및 텔로미어의 유지와 같은 과정에서 매우 중요하다.[57] 세포 내에서 이러한 기능 외에도 최근에 발견된 세포 외 ADP-리보실트랜스퍼레이스는 그 기능이 아직 불명확하다.[58] NAD+는 또한 5' 말단 변형으로 세포질의 RNA 상에 첨가될 수 있다.[59]
세포 신호전달에서 NAD+의 또 다른 기능은 2차 신호전달 시스템의 일부로 ADP-리보실 고리화효소에 의해 NAD+로부터 생성되는 고리형 ADP-리보스의 전구물질로 사용되는 것이다.[60] 고리형 ADP-리보스는 세포 내 저장소로부터 칼슘을 방출하게 함으로써 칼슘 신호전달에 작용한다.[61] 고리형 ADP-리보스는 소포체와 같은 세포소기관의 막에 위치한 리아노딘 수용체라고 불리는 칼슘 통로에 결합하고 칼슘 통로를 열리게 한다.[62]
또한 NAD+는 Sir2와 같은 NAD-의존성 탈아세틸화효소인 시르투인에 의해 소모된다.[63] 이 효소는 아세틸기를 기질 단백질로부터 NAD+의 ADP-리보스 잔기로 전달함으로써 작용한다. NAD-의존성 탈아세틸화효소는 조효소를 분해하고, 니코틴아마이드와 O-아세틸-ADP-리보스를 방출한다. 시르투인은 주로 히스톤의 탈아세틸화와 뉴클레오솜의 구조를 바꾸는 것을 통해 전사 조절에 관여하는 것으로 보인다.[64] 또한 비히스톤 단백질은 시르투인에 의해서도 탈아세틸화될 수 있다. 이러한 시르투인의 활동은 노화 조절에 있어서 중요하기 때문에 특히 흥미롭다.[65]
다른 NAD-의존성 효소에는 하나의 DNA 말단의 5' 인산기에 아데노신 일인산(AMP) 잔기를 공여하기 위한 기질로써 NAD+를 사용하여 두 DNA 말단을 결합시키는 세균의 DNA 리게이스가 있다. 이러한 중간생성물은 다른 DNA 말단의 3' 하이드록시기에 의해 공격을 받아 새로운 포스포다이에스터 결합을 형성한다.[66] 이것은 ATP를 사용하여 DNA-AMP 중간생성물을 형성하는 진핵생물의 DNA 리게이스와는 대조적이다.[67]
세포 외에서 NAD+의 작용
편집최근 몇 년간 NAD+는 세포와 세포 간의 신호전달에 관여하는 세포 외 신호전달 분자로 인식되고 있다.[36][68][69] NAD+는 혈관,[35] 방광,[35][70] 대장[71][72]의 뉴런으로부터 방출되고, 신경분비세포[73] 및 뇌의 시냅토솜[74]으로부터 방출되며, 신경으로부터 평활근의 반응기 세포로 정보를 전달하는 새로운 신경전달물질로 제안되고 있다.[71][72] NAD+의 세포 외 작용에 대한 기본 메커니즘 및 사람의 건강과 질병에 대한 중요성을 결정하기 위해서는 더 많은 연구가 필요하다.
연구
편집NAD+와 NADH를 만들고 사용하는 효소는 약리학과 질병에 대한 치료법 연구 모두에서 중요하다.[75] 약물 디자인과 약물 개발은 NAD+를 약물의 직접적인 표적으로 NAD-의존성 효소들의 활성을 변화시키는 구조를 바탕으로 효소 저해제나 효소 활성제를 설계하는 것, NAD+ 생합성의 억제를 시도하는 것과 같은 방법으로 이용한다.[76]
NAD+는 알츠하이머병 및 파킨슨병과 같은 신경퇴행성 질환의 치료에 잠재적인 이용을 위해 연구되어 왔다.[2] 파킨슨병 환자들을 대상으로 위약 조절 임상시험은 어떤 효과도 보여주지 못했다.[77]
NAD+는 결핵균에 의한 감염성 질병인 결핵의 치료에 사용되는 항결핵성 항생제인 아이소나이아지드의 직접적인 표적이기도 하다. 아이소나이아지드는 프로드러그이며, 일단 세균 속으로 침투하면, 과산화효소에 의해 활성화되어 화합물을 자유 라디칼 형태로 산화시킨다.[78] 이 라디칼은 NADH와 반응하여 에노일-아실-운반체 단백질 환원효소[79] 및 다이하이드로폴산 환원효소에 대한 매우 강력한 저해제를 생성한다.[80]
다수의 산화환원효소들이 NAD+와 NADH를 기질로 사용하고, 고도로 보존된 구조 모티프를 사용하여 결합하기 때문에, NAD+에 기초한 저해제가 하나의 효소에 특이적일 수 있다는 생각은 놀랍다.[81] 예를 들어 마이코페놀산 및 티아조푸린에 기초한 저해제는 NAD+ 결합 부위에서 IMP 탈수소효소를 억제한다. 퓨린 대사에서 IMP 탈수소효소의 중요성 때문에 이들 화합물은 항암제, 항바이러스제 또는 면역억제제로 유용할 수 있다.[81][82] 다른 약물들은 효소 저해제가 아니라, NAD+ 대사에 관여하는 효소를 활성화시킨다. 이러한 NAD-의존성 탈아세틸화효소의 활성화가 일부 동물에서 수명을 연장시키는 것으로 확인되었기 때문에 그러한 약물들에게 시르투인은 특히 관심 표적이다.[83] 레스베라트롤과 같은 화합물은 이러한 효소들의 활성을 증가시키는데, 이는 척추동물[84]과 무척추동물에서 노화를 지연시키는 능력에 중요할 수 있다.[85][86] 한 실험에서 1주 동안 NAD를 투여받은 쥐들은 세포핵과 미토콘드리아 간의 연락이 향상되었다.[87]
세균과 사람 간의 대사 경로의 차이처럼 생물체 간의 NAD+ 생합성 대사 경로의 차이 때문에 이러한 물질대사의 영역은 새로운 항생제 개발을 위한 유망한 영역이다.[88][89] 예를 들어 니코틴아마이드를 니코틴산으로 전환시키는 효소인 니코틴아미데이스는 사람에게는 존재하지 않지만, 효모 및 세균에 존재하기 때문에 약물 디자인을 위한 대상이 된다.[30]
세균학에서 때로는 인자 V(factor V)로 언급되는 NAD는 몇몇 까다로운 세균 배지에 보충제로 사용된다.[90]
역사
편집조효소 NAD+는 1906년 영국의 생화학자인 아서 하든과 윌리엄 존 영에 의해 처음 발견되었다.[91] 그들은 끓이지 않은 효모 추출액에 끓이고 여과된 효모 추출액을 첨가하면 알코올 발효가 크게 촉진된다는 것을 알게 되었다. 그들은 이러한 효과의 원인이 되는 미확인 요인을 "coferment"(조효소(coenzyme)의 옛 명칭) 라고 불렀다. 효모 추출액으로부터 길고 어려운 정제 과정을 통해, 이 열에 안정한 인자는 한스 폰 오일러켈핀에 의해 당뉴클레오타이드 인산으로 확인되었다.[92] 1936년 독일의 과학자 오토 하인리히 바르부르크는 하이드라이드의 전달에서 뉴클레오타이드 조효소의 기능을 보여주었고, 니코틴아마이드 부분을 산화환원반응의 부위로 확인했다.[93]
NAD+의 비타민 전구물질은 1938년에 콘래드 엘베예른(Conrad Elvehjem)이 간이 니코틴아마이드 형태의 "anti-black tongue" 활성을 갖는다는 것을 보여줌으로 인해 처음 확인되었다.[94] 1939년에 엘베예른은 니아신이 NAD+ 합성에 사용된다는 최초의 강력한 증거를 제시했다.[95] 1940년대 초 아서 콘버그는 생합성 경로에서 NAD+ 대사를 이해하는데 중요한 기여를 했다.[96] 1949년에 미국의 생화학자인 모리스 프리드킨(Morris Friedkin)과 앨버트 L. 레닌저는 NADH가 시트르산 회로와 같은 대사 경로를 산화적 인산화의 ATP 합성과 연계시킨다는 것을 증명했다.[97] 1958년에 잭 프레이스(Jack Preiss)와 필립 핸들러(Philip Handler)는 NAD+의 생합성에 관여하는 중간생성물과 효소를 발견했다.[98][99] 니코틴산으로부터 회수 합성은 프레이스-핸들러(Preiss-Handler) 경로라고 불린다. 2004년에 찰스 브레너(Charles Brenner)와 동료들은 NAD+로 가는 니코틴아마이드 리보사이드 키네이스 경로를 발견했다.[100] 니코틴아마이드 리보사이드는 현재 "Tru Niagen"이라는 브랜드명으로 크로마덱스사(社)에 의해 제조되고 있다.
NAD(P)의 비산화환원반응에서의 역할은 나중에 발견되었다.[1] 가장 먼저 확인된 것은 1960년대 초에 관찰된 ADP-리보실화 반응에서 NAD+를 ADP-리보스 공여체로 사용하는 것이었다.[101] 1980년대와 1990년대의 연구에서 1987년에 발견된 고리형 ADP-리보스의 작용과 같은 세포 신호전달에서 NAD+와 NADP+ 대사산물의 활성이 밝혀졌다.[102] NAD+의 대사는 2000년에 이마이 신이치로(今井眞一郞, Imai Shin-ichiro)와 동료들에 의해 시르투인이라고 불리는 NAD+-의존성 단백질 탈아세틸화효소가 발견된 후로 관심이 높아지는 등 21세기에도 치열한 연구 영역으로 남아있다.[103]
같이 보기
편집각주
편집- ↑ 가 나 다 Pollak N, Dölle C, Ziegler M (2007). “The power to reduce: pyridine nucleotides – small molecules with a multitude of functions”. 《Biochem. J.》 402 (2): 205–18. doi:10.1042/BJ20061638. PMC 1798440. PMID 17295611.
- ↑ 가 나 다 라 마 바 Belenky P, Bogan KL, Brenner C (2007). “NAD+ metabolism in health and disease” (PDF). 《Trends Biochem. Sci.》 32 (1): 12–9. doi:10.1016/j.tibs.2006.11.006. PMID 17161604. 4 July 2009에 원본 문서 (PDF)에서 보존된 문서. 23 December 2007에 확인함.
- ↑ Unden G, Bongaerts J (1997). “Alternative respiratory pathways of Escherichia coli: energetics and transcriptional regulation in response to electron acceptors”. 《Biochim. Biophys. Acta》 1320 (3): 217–34. doi:10.1016/S0005-2728(97)00034-0. PMID 9230919.
- ↑ Windholz, Martha (1983). 《The Merck Index: an encyclopedia of chemicals, drugs, and biologicals》 10판. Rahway NJ, US: Merck. 909쪽. ISBN 0-911910-27-1.
- ↑ Biellmann JF, Lapinte C, Haid E, Weimann G (1979). “Structure of lactate dehydrogenase inhibitor generated from coenzyme”. 《Biochemistry》 18 (7): 1212–7. doi:10.1021/bi00574a015. PMID 218616.
- ↑ 가 나 Dawson, R. Ben (1985). 《Data for biochemical research》 3판. Oxford: Clarendon Press. 122쪽. ISBN 0-19-855358-7.
- ↑ 가 나 Lakowicz JR, Szmacinski H, Nowaczyk K, Johnson ML (1992). “Fluorescence lifetime imaging of free and protein-bound NADH”. 《Proc. Natl. Acad. Sci. U.S.A.》 89 (4): 1271–5. Bibcode:1992PNAS...89.1271L. doi:10.1073/pnas.89.4.1271. PMC 48431. PMID 1741380.
- ↑ Jameson DM, Thomas V, Zhou DM (1989). “Time-resolved fluorescence studies on NADH bound to mitochondrial malate dehydrogenase”. 《Biochim. Biophys. Acta》 994 (2): 187–90. doi:10.1016/0167-4838(89)90159-3. PMID 2910350.
- ↑ Kasimova MR, Grigiene J, Krab K, Hagedorn PH, Flyvbjerg H, Andersen PE, Møller IM (2006). “The Free NADH Concentration Is Kept Constant in Plant Mitochondria under Different Metabolic Conditions”. 《Plant Cell》 18 (3): 688–98. doi:10.1105/tpc.105.039354. PMC 1383643. PMID 16461578.
- ↑ Reiss PD, Zuurendonk PF, Veech RL (1984). “Measurement of tissue purine, pyrimidine, and other nucleotides by radial compression high-performance liquid chromatography”. 《Anal. Biochem.》 140 (1): 162–71. doi:10.1016/0003-2697(84)90148-9. PMID 6486402.
- ↑ Yamada K, Hara N, Shibata T, Osago H, Tsuchiya M (2006). “The simultaneous measurement of nicotinamide adenine dinucleotide and related compounds by liquid chromatography/electrospray ionization tandem mass spectrometry”. 《Anal. Biochem.》 352 (2): 282–5. doi:10.1016/j.ab.2006.02.017. PMID 16574057.
- ↑ 가 나 Yang H, Yang T, Baur JA, Perez E, Matsui T, Carmona JJ, Lamming DW, Souza-Pinto NC, Bohr VA, Rosenzweig A, de Cabo R, Sauve AA, Sinclair DA (2007). “Nutrient-Sensitive Mitochondrial NAD+ Levels Dictate Cell Survival”. 《Cell》 130 (6): 1095–107. doi:10.1016/j.cell.2007.07.035. PMC 3366687. PMID 17889652.
- ↑ Belenky P, Racette FG, Bogan KL, McClure JM, Smith JS, Brenner C (2007). “Nicotinamide riboside promotes Sir2 silencing and extends lifespan via Nrk and Urh1/Pnp1/Meu1 pathways to NAD+”. 《Cell》 129 (3): 473–84. doi:10.1016/j.cell.2007.03.024. PMID 17482543.
- ↑ Blinova K, Carroll S, Bose S, Smirnov AV, Harvey JJ, Knutson JR, Balaban RS (2005). “Distribution of mitochondrial NADH fluorescence lifetimes: steady-state kinetics of matrix NADH interactions”. 《Biochemistry》 44 (7): 2585–94. doi:10.1021/bi0485124. PMID 15709771.
- ↑ Todisco S, Agrimi G, Castegna A, Palmieri F (2006). “Identification of the mitochondrial NAD+ transporter in Saccharomyces cerevisiae”. 《J. Biol. Chem.》 281 (3): 1524–31. doi:10.1074/jbc.M510425200. PMID 16291748.
- ↑ Schafer FQ, Buettner GR (2001). “Redox environment of the cell as viewed through the redox state of the glutathione disulfide/glutathione couple”. 《Free Radic Biol Med》 30 (11): 1191–212. doi:10.1016/S0891-5849(01)00480-4. PMID 11368918.
- ↑ Williamson DH, Lund P, Krebs HA (1967). “The redox state of free nicotinamide-adenine dinucleotide in the cytoplasm and mitochondria of rat liver”. 《Biochem. J.》 103 (2): 514–27. PMC 1270436. PMID 4291787.
- ↑ Zhang Q, Piston DW, Goodman RH (2002). “Regulation of corepressor function by nuclear NADH”. 《Science》 295 (5561): 1895–7. doi:10.1126/science.1069300. PMID 11847309.
- ↑ Lin SJ, Guarente L (April 2003). “Nicotinamide adenine dinucleotide, a metabolic regulator of transcription, longevity and disease”. 《Curr. Opin. Cell Biol.》 15 (2): 241–6. doi:10.1016/S0955-0674(03)00006-1. PMID 12648681.
- ↑ Veech RL, Eggleston LV, Krebs HA (1969). “The redox state of free nicotinamide–adenine dinucleotide phosphate in the cytoplasm of rat liver”. 《Biochem. J.》 115 (4): 609–19. doi:10.1042/bj1150609a. PMC 1185185. PMID 4391039.
- ↑ Katoh A, Uenohara K, Akita M, Hashimoto T (2006). “Early Steps in the Biosynthesis of NAD in Arabidopsis Start with Aspartate and Occur in the Plastid”. 《Plant Physiol.》 141 (3): 851–7. doi:10.1104/pp.106.081091. PMC 1489895. PMID 16698895.
- ↑ Foster JW, Moat AG (1980년 3월 1일). “Nicotinamide adenine dinucleotide biosynthesis and pyridine nucleotide cycle metabolism in microbial systems”. 《Microbiol. Rev.》 44 (1): 83–105. PMC 373235. PMID 6997723.
- ↑ Magni G, Orsomando G, Raffaelli N (2006). “Structural and functional properties of NAD kinase, a key enzyme in NADP biosynthesis”. 《Mini Reviews in Medicinal Chemistry》 6 (7): 739–46. doi:10.2174/138955706777698688. PMID 16842123.
- ↑ Sakuraba H, Kawakami R, Ohshima T (2005). “First Archaeal Inorganic Polyphosphate/ATP-Dependent NAD Kinase, from Hyperthermophilic Archaeon Pyrococcus horikoshii: Cloning, Expression, and Characterization”. 《Appl. Environ. Microbiol.》 71 (8): 4352–8. doi:10.1128/AEM.71.8.4352-4358.2005. PMC 1183369. PMID 16085824.
- ↑ Raffaelli N, Finaurini L, Mazzola F, Pucci L, Sorci L, Amici A, Magni G (2004). “Characterization of Mycobacterium tuberculosis NAD kinase: functional analysis of the full-length enzyme by site-directed mutagenesis”. 《Biochemistry》 43 (23): 7610–7. doi:10.1021/bi049650w. PMID 15182203.
- ↑ Anderson RM, Bitterman KJ, Wood JG, Medvedik O, Cohen H, Lin SS, Manchester JK, Gordon JI, Sinclair DA (2002). “Manipulation of a nuclear NAD+ salvage pathway delays aging without altering steady-state NAD+ levels”. 《J. Biol. Chem.》 277 (21): 18881–90. doi:10.1074/jbc.M111773200. PMC 3745358. PMID 11884393.
- ↑ Billington RA, Travelli C, Ercolano E, Galli U, Roman CB, Grolla AA, Canonico PL, Condorelli F, Genazzani AA (2008). “Characterization of NAD Uptake in Mammalian Cells”. 《J. Biol. Chem.》 283 (10): 6367–74. doi:10.1074/jbc.M706204200. PMID 18180302.
- ↑ Trammell SA, Schmidt MS, Weidemann BJ, Redpath P, Jaksch F, Dellinger RW, Li Z, Abel ED, Migaud ME, Brenner C (2016). “Nicotinamide riboside is uniquely and orally bioavailable in mice and humans”. 《Nature Communications》 7: 12948. doi:10.1038/ncomms12948. PMID 27721479.
- ↑ Henderson LM (1983). “Niacin”. 《Annu. Rev. Nutr.》 3: 289–307. doi:10.1146/annurev.nu.03.070183.001445. PMID 6357238.
- ↑ 가 나 Rongvaux A, Andris F, Van Gool F, Leo O (2003). “Reconstructing eukaryotic NAD metabolism”. 《BioEssays》 25 (7): 683–90. doi:10.1002/bies.10297. PMID 12815723.
- ↑ Ma B, Pan SJ, Zupancic ML, Cormack BP (2007). “Assimilation of NAD+ precursors in Candida glabrata”. 《Mol. Microbiol.》 66 (1): 14–25. doi:10.1111/j.1365-2958.2007.05886.x. PMID 17725566.
- ↑ Reidl J, Schlör S, Kraiss A, Schmidt-Brauns J, Kemmer G, Soleva E (2000). “NADP and NAD utilization in Haemophilus influenzae”. 《Mol. Microbiol.》 35 (6): 1573–81. doi:10.1046/j.1365-2958.2000.01829.x. PMID 10760156.
- ↑ Gerdes SY, Scholle MD, D'Souza M, Bernal A, Baev MV, Farrell M, Kurnasov OV, Daugherty MD, Mseeh F, Polanuyer BM, Campbell JW, Anantha S, Shatalin KY, Chowdhury SA, Fonstein MY, Osterman AL (2002). “From Genetic Footprinting to Antimicrobial Drug Targets: Examples in Cofactor Biosynthetic Pathways”. 《J. Bacteriol.》 184 (16): 4555–72. doi:10.1128/JB.184.16.4555-4572.2002. PMC 135229. PMID 12142426.
- ↑ Senkovich O, Speed H, Grigorian A, 외. (2005). “Crystallization of three key glycolytic enzymes of the opportunistic pathogen Cryptosporidium parvum”. 《Biochim. Biophys. Acta》 1750 (2): 166–72. doi:10.1016/j.bbapap.2005.04.009. PMID 15953771.
- ↑ 가 나 다 Smyth LM, Bobalova J, Mendoza MG, Lew C, Mutafova-Yambolieva VN (2004). “Release of beta-nicotinamide adenine dinucleotide upon stimulation of postganglionic nerve terminals in blood vessels and urinary bladder”. 《J Biol Chem》 279 (47): 48893–903. doi:10.1074/jbc.M407266200. PMID 15364945.
- ↑ 가 나 다 Billington RA, Bruzzone S, De Flora A, Genazzani AA, Koch-Nolte F, Ziegler M, Zocchi E (2006). “Emerging functions of extracellular pyridine nucleotides”. 《Mol. Med.》 12 (11–12): 324–7. doi:10.2119/2006-00075.Billington. PMC 1829198. PMID 17380199.
- ↑ “Enzyme Nomenclature, Recommendations for enzyme names from the Nomenclature Committee of the International Union of Biochemistry and Molecular Biology”. 5 December 2007에 원본 문서에서 보존된 문서. 6 December 2007에 확인함.
- ↑ “NiceZyme View of ENZYME: EC 1.6.5.3”. Expasy. 2007년 12월 16일에 확인함.
- ↑ Hanukoglu I (2015). “Proteopedia: Rossmann fold: A beta-alpha-beta fold at dinucleotide binding sites”. 《Biochem Mol Biol Educ》 43 (3): 206–209. doi:10.1002/bmb.20849. PMID 25704928.
- ↑ Lesk AM (1995). “NAD-binding domains of dehydrogenases”. 《Curr. Opin. Struct. Biol.》 5 (6): 775–83. doi:10.1016/0959-440X(95)80010-7. PMID 8749365.
- ↑ Rao ST, Rossmann MG (1973). “Comparison of super-secondary structures in proteins”. 《J Mol Biol》 76 (2): 241–56. doi:10.1016/0022-2836(73)90388-4. PMID 4737475.
- ↑ Goto M, Muramatsu H, Mihara H, Kurihara T, Esaki N, Omi R, Miyahara I, Hirotsu K (2005). “Crystal structures of Delta1-piperideine-2-carboxylate/Delta1-pyrroline-2-carboxylate reductase belonging to a new family of NAD(P)H-dependent oxidoreductases: conformational change, substrate recognition, and stereochemistry of the reaction”. 《J. Biol. Chem.》 280 (49): 40875–84. doi:10.1074/jbc.M507399200. PMID 16192274.
- ↑ 가 나 Bellamacina CR (1996년 9월 1일). “The nicotinamide dinucleotide binding motif: a comparison of nucleotide binding proteins”. 《FASEB J.》 10 (11): 1257–69. PMID 8836039.
- ↑ Carugo O, Argos P (1997). “NADP-dependent enzymes. I: Conserved stereochemistry of cofactor binding”. 《Proteins》 28 (1): 10–28. doi:10.1002/(SICI)1097-0134(199705)28:1<10::AID-PROT2>3.0.CO;2-N. PMID 9144787.
- ↑ Vickers TJ, Orsomando G, de la Garza RD, Scott DA, Kang SO, Hanson AD, Beverley SM (2006). “Biochemical and genetic analysis of methylenetetrahydrofolate reductase in Leishmania metabolism and virulence”. 《J. Biol. Chem.》 281 (50): 38150–8. doi:10.1074/jbc.M608387200. PMID 17032644.
- ↑ Bakker BM, Overkamp KM, Kötter P, Luttik MA, Pronk JT (2001). “Stoichiometry and compartmentation of NADH metabolism in Saccharomyces cerevisiae”. 《FEMS Microbiol. Rev.》 25 (1): 15–37. doi:10.1111/j.1574-6976.2001.tb00570.x. PMID 11152939.
- ↑ Rich PR (2003). “The molecular machinery of Keilin's respiratory chain”. 《Biochem. Soc. Trans.》 31 (Pt 6): 1095–105. doi:10.1042/BST0311095. PMID 14641005.
- ↑ Heineke D, Riens B, Grosse H, Hoferichter P, Peter U, Flügge UI, Heldt HW (1991). “Redox Transfer across the Inner Chloroplast Envelope Membrane”. 《Plant Physiol》 95 (4): 1131–1137. doi:10.1104/pp.95.4.1131. PMC 1077662. PMID 16668101.
- ↑ 가 나 Nicholls DG; Ferguson SJ (2002). 《Bioenergetics 3》 1판. Academic Press. ISBN 0-12-518121-3.
- ↑ Sistare FD, Haynes RC (1985년 10월 15일). “The interaction between the cytosolic pyridine nucleotide redox potential and gluconeogenesis from lactate/pyruvate in isolated rat hepatocytes. Implications for investigations of hormone action”. 《J. Biol. Chem.》 260 (23): 12748–53. PMID 4044607. 2008년 4월 12일에 원본 문서에서 보존된 문서. 2019년 2월 18일에 확인함.
- ↑ Freitag A, Bock E (1990). “Energy conservation in Nitrobacter”. 《FEMS Microbiology Letters》 66 (1–3): 157–62. doi:10.1111/j.1574-6968.1990.tb03989.x.
- ↑ Starkenburg SR, Chain PS, Sayavedra-Soto LA, Hauser L, Land ML, Larimer FW, Malfatti SA, Klotz MG, Bottomley PJ, Arp DJ, Hickey WJ (2006). “Genome Sequence of the Chemolithoautotrophic Nitrite-Oxidizing Bacterium Nitrobacter winogradskyi Nb-255”. 《Appl. Environ. Microbiol.》 72 (3): 2050–63. doi:10.1128/AEM.72.3.2050-2063.2006. PMC 1393235. PMID 16517654. 2019년 3월 5일에 원본 문서에서 보존된 문서. 2019년 2월 18일에 확인함.
- ↑ Ziegler M (2000). “New functions of a long-known molecule. Emerging roles of NAD in cellular signaling”. 《Eur. J. Biochem.》 267 (6): 1550–64. doi:10.1046/j.1432-1327.2000.01187.x. PMID 10712584.
- ↑ 가 나 Diefenbach J, Bürkle A (2005). “Introduction to poly(ADP-ribose) metabolism”. 《Cell. Mol. Life Sci.》 62 (7–8): 721–30. doi:10.1007/s00018-004-4503-3. PMID 15868397.
- ↑ Berger F, Ramírez-Hernández MH, Ziegler M (2004). “The new life of a centenarian: signaling functions of NAD(P)”. 《Trends Biochem. Sci.》 29 (3): 111–8. doi:10.1016/j.tibs.2004.01.007. PMID 15003268.
- ↑ Corda D, Di Girolamo M (2003). “New Embo Member's Review: Functional aspects of protein mono-ADP-ribosylation”. 《EMBO J.》 22 (9): 1953–8. doi:10.1093/emboj/cdg209. PMC 156081. PMID 12727863.
- ↑ 가 나 Bürkle A (2005). “Poly(ADP-ribose). The most elaborate metabolite of NAD+”. 《FEBS J.》 272 (18): 4576–89. doi:10.1111/j.1742-4658.2005.04864.x. PMID 16156780.
- ↑ Seman M, Adriouch S, Haag F, Koch-Nolte F (2004). “Ecto-ADP-ribosyltransferases (ARTs): emerging actors in cell communication and signaling”. 《Curr. Med. Chem.》 11 (7): 857–72. doi:10.2174/0929867043455611. PMID 15078170.
- ↑ Chen YG, Kowtoniuk WE, Agarwal I, Shen Y, Liu DR (December 2009). “LC/MS analysis of cellular RNA reveals NAD-linked RNA”. 《Nat Chem Biol》 5 (12): 879–881. doi:10.1038/nchembio.235. PMC 2842606. PMID 19820715.
- ↑ Guse AH (2004). “Biochemistry, biology, and pharmacology of cyclic adenosine diphosphoribose (cADPR)”. 《Curr. Med. Chem.》 11 (7): 847–55. doi:10.2174/0929867043455602. PMID 15078169.
- ↑ Guse AH (2004). “Regulation of calcium signaling by the second messenger cyclic adenosine diphosphoribose (cADPR)”. 《Curr. Mol. Med.》 4 (3): 239–48. doi:10.2174/1566524043360771. PMID 15101682.
- ↑ Guse AH (2005). “Second messenger function and the structure-activity relationship of cyclic adenosine diphosphoribose (cADPR)”. 《FEBS J.》 272 (18): 4590–7. doi:10.1111/j.1742-4658.2005.04863.x. PMID 16156781.
- ↑ North BJ, Verdin E (2004). “Sirtuins: Sir2-related NAD-dependent protein deacetylases”. 《Genome Biol》 5 (5): 224. doi:10.1186/gb-2004-5-5-224. PMC 416462. PMID 15128440.
- ↑ Blander G, Guarente L (2004). “The Sir2 family of protein deacetylases”. 《Annu. Rev. Biochem.》 73: 417–35. doi:10.1146/annurev.biochem.73.011303.073651. PMID 15189148.
- ↑ Trapp J, Jung M (2006). “The role of NAD+ dependent histone deacetylases (sirtuins) in ageing”. 《Curr Drug Targets》 7 (11): 1553–60. doi:10.2174/1389450110607011553. PMID 17100594.
- ↑ Wilkinson A, Day J, Bowater R (2001). “Bacterial DNA ligases”. 《Mol. Microbiol.》 40 (6): 1241–8. doi:10.1046/j.1365-2958.2001.02479.x. PMID 11442824.
- ↑ Schär P, Herrmann G, Daly G, Lindahl T (1997). “A newly identified DNA ligase of Saccharomyces cerevisiae involved in RAD52-independent repair of DNA double-strand breaks”. 《Genes & Development》 11 (15): 1912–24. doi:10.1101/gad.11.15.1912. PMC 316416. PMID 9271115.
- ↑ Ziegler M, Niere M (2004). “NAD+ surfaces again”. 《Biochem. J.》 382 (Pt 3): e5–6. doi:10.1042/BJ20041217. PMC 1133982. PMID 15352307.
- ↑ Koch-Nolte F, Fischer S, Haag F, Ziegler M (2011). “Compartmentation of NAD+-dependent signalling”. 《FEBS Lett.》 585 (11): 1651–6. doi:10.1016/j.febslet.2011.03.045. PMID 21443875.
- ↑ Breen LT, Smyth LM, Yamboliev IA, Mutafova-Yambolieva VN (2006). “beta-NAD is a novel nucleotide released on stimulation of nerve terminals in human urinary bladder detrusor muscle”. 《Am. J. Physiol. Renal Physiol.》 290 (2): F486–95. doi:10.1152/ajprenal.00314.2005. PMID 16189287.
- ↑ 가 나 Mutafova-Yambolieva VN, Hwang SJ, Hao X, Chen H, Zhu MX, Wood JD, Ward SM, Sanders KM (2007). “Beta-nicotinamide adenine dinucleotide is an inhibitory neurotransmitter in visceral smooth muscle”. 《Proc. Natl. Acad. Sci. U.S.A.》 104 (41): 16359–64. Bibcode:2007PNAS..10416359M. doi:10.1073/pnas.0705510104. PMC 2042211. PMID 17913880.
- ↑ 가 나 Hwang SJ, Durnin L, Dwyer L, Rhee PL, Ward SM, Koh SD, Sanders KM, Mutafova-Yambolieva VN (2011). “β-nicotinamide adenine dinucleotide is an enteric inhibitory neurotransmitter in human and nonhuman primate colons”. 《Gastroenterology》 140 (2): 608–617.e6. doi:10.1053/j.gastro.2010.09.039. PMC 3031738. PMID 20875415.
- ↑ Yamboliev IA, Smyth LM, Durnin L, Dai Y, Mutafova-Yambolieva VN (2009). “Storage and secretion of beta-NAD, ATP and dopamine in NGF-differentiated rat pheochromocytoma PC12 cells”. 《Eur. J. Neurosci.》 30 (5): 756–68. doi:10.1111/j.1460-9568.2009.06869.x. PMC 2774892. PMID 19712094.
- ↑ Durnin L, Dai Y, Aiba I, Shuttleworth CW, Yamboliev IA, Mutafova-Yambolieva VN (2012). “Release, neuronal effects and removal of extracellular β-nicotinamide adenine dinucleotide (β-NAD+) in the rat brain”. 《Eur. J. Neurosci.》 35 (3): 423–35. doi:10.1111/j.1460-9568.2011.07957.x. PMC 3270379. PMID 22276961.
- ↑ Sauve AA (March 2008). “NAD+ and vitamin B3: from metabolism to therapies”. 《The Journal of Pharmacology and Experimental Therapeutics》 324 (3): 883–93. doi:10.1124/jpet.107.120758. PMID 18165311.
- ↑ Khan JA, Forouhar F, Tao X, Tong L (2007). “Nicotinamide adenine dinucleotide metabolism as an attractive target for drug discovery”. 《Expert Opin. Ther. Targets》 11 (5): 695–705. doi:10.1517/14728222.11.5.695. PMID 17465726.
- ↑ Swerdlow RH (1998). “Is NADH effective in the treatment of Parkinson's disease?”. 《Drugs Aging》 13 (4): 263–8. doi:10.2165/00002512-199813040-00002. PMID 9805207.
- ↑ Timmins GS, Deretic V (2006). “Mechanisms of action of isoniazid”. 《Mol. Microbiol.》 62 (5): 1220–7. doi:10.1111/j.1365-2958.2006.05467.x. PMID 17074073.
- ↑ Rawat R, Whitty A, Tonge PJ (2003). “The isoniazid-NAD adduct is a slow, tight-binding inhibitor of InhA, the Mycobacterium tuberculosis enoyl reductase: Adduct affinity and drug resistance”. 《Proc. Natl. Acad. Sci. U.S.A.》 100 (24): 13881–6. Bibcode:2003PNAS..10013881R. doi:10.1073/pnas.2235848100. PMC 283515. PMID 14623976.
- ↑ Argyrou A, Vetting MW, Aladegbami B, Blanchard JS (2006). “Mycobacterium tuberculosis dihydrofolate reductase is a target for isoniazid”. 《Nat. Struct. Mol. Biol.》 13 (5): 408–13. doi:10.1038/nsmb1089. PMID 16648861.
- ↑ 가 나 Pankiewicz KW, Patterson SE, Black PL, Jayaram HN, Risal D, Goldstein BM, Stuyver LJ, Schinazi RF (2004). “Cofactor mimics as selective inhibitors of NAD-dependent inosine monophosphate dehydrogenase (IMPDH)—the major therapeutic target”. 《Curr. Med. Chem.》 11 (7): 887–900. doi:10.2174/0929867043455648. PMID 15083807.
- ↑ Franchetti P, Grifantini M (1999). “Nucleoside and non-nucleoside IMP dehydrogenase inhibitors as antitumor and antiviral agents”. 《Curr. Med. Chem.》 6 (7): 599–614. PMID 10390603.
- ↑ Kim EJ, Um SJ (2008). “SIRT1: roles in aging and cancer”. 《BMB Rep》 41 (11): 751–6. doi:10.5483/BMBRep.2008.41.11.751. PMID 19017485.
- ↑ Valenzano DR, Terzibasi E, Genade T, Cattaneo A, Domenici L, Cellerino A (2006). “Resveratrol prolongs lifespan and retards the onset of age-related markers in a short-lived vertebrate”. 《Curr. Biol.》 16 (3): 296–300. doi:10.1016/j.cub.2005.12.038. PMID 16461283.
- ↑ Howitz KT, Bitterman KJ, Cohen HY, Lamming DW, Lavu S, Wood JG, Zipkin RE, Chung P, Kisielewski A, Zhang LL, Scherer B, Sinclair DA (2003). “Small molecule activators of sirtuins extend Saccharomyces cerevisiae lifespan”. 《Nature》 425 (6954): 191–6. Bibcode:2003Natur.425..191H. doi:10.1038/nature01960. PMID 12939617.
- ↑ Wood JG, Rogina B, Lavu S, Howitz K, Helfand SL, Tatar M, Sinclair D (2004). “Sirtuin activators mimic caloric restriction and delay ageing in metazoans”. 《Nature》 430 (7000): 686–9. Bibcode:2004Natur.430..686W. doi:10.1038/nature02789. PMID 15254550.
- ↑ Gomes AP, Price NL, Ling AJ, Moslehi JJ, Montgomery MK, Rajman L, White JP, Teodoro JS, Wrann CD, Hubbard BP, Mercken EM, Palmeira CM, de Cabo R, Rolo AP, Turner N, Bell EL, Sinclair DA (2013년 12월 19일). “Declining NAD+ Induces a Pseudohypoxic State Disrupting Nuclear-Mitochondrial Communication during Aging”. 《Cell》 155 (7): 1624–1638. doi:10.1016/j.cell.2013.11.037. PMC 4076149. PMID 24360282.
- ↑ Rizzi M, Schindelin H (2002). “Structural biology of enzymes involved in NAD and molybdenum cofactor biosynthesis”. 《Curr. Opin. Struct. Biol.》 12 (6): 709–20. doi:10.1016/S0959-440X(02)00385-8. PMID 12504674.
- ↑ Begley TP, Kinsland C, Mehl RA, Osterman A, Dorrestein P (2001). “The biosynthesis of nicotinamide adenine dinucleotides in bacteria”. 《Vitam. Horm.》. Vitamins & Hormones 61: 103–19. doi:10.1016/S0083-6729(01)61003-3. ISBN 978-0-12-709861-6. PMID 11153263.
- ↑ Meningitis |Lab Manual |Id and Characterization of Hib |CDC
- ↑ Harden, A; Young, WJ (1906년 10월 24일). “The alcoholic ferment of yeast-juice Part II.--The coferment of yeast-juice”. 《Proceedings of the Royal Society of London》. Series B, Containing Papers of a Biological Character 78 (526): 369–375. doi:10.1098/rspb.1906.0070. JSTOR 80144.
- ↑ “Fermentation of sugars and fermentative enzymes” (PDF). 《Nobel Lecture, 23 May 1930》. Nobel Foundation. 27 September 2007에 원본 문서 (PDF)에서 보존된 문서. 30 September 2007에 확인함.
- ↑ Warburg O, Christian W (1936). “Pyridin, der wasserstoffübertragende bestandteil von gärungsfermenten (pyridin-nucleotide)” [Pyridin, the hydrogen-transferring component of the fermentation enzymes (pyridine nucleotide)]. 《Biochemische Zeitschrift》 (독일어) 287: 291. doi:10.1002/hlca.193601901199.
- ↑ Elvehjem CA, Madden RJ, Strong FM, Woolley DW (1938). “The isolation and identification of the anti-black tongue factor” (PDF). 《J. Biol. Chem.》 123 (1): 137–49. 2009년 3월 26일에 원본 문서 (PDF)에서 보존된 문서. 2019년 2월 18일에 확인함.
- ↑ Axelrod AE, Madden RJ, Elvehjem CA (1939). “The effect of a nicotinic acid deficiency upon the coenzyme I content of animal tissues” (PDF). 《J. Biol. Chem.》 131 (1): 85–93. 2009년 3월 26일에 원본 문서 (PDF)에서 보존된 문서. 2019년 2월 18일에 확인함.
- ↑ Kornberg A (1948). “The participation of inorganic pyrophosphate in the reversible enzymatic synthesis of diphosphopyridine nucleotide” (PDF). 《J. Biol. Chem.》 176 (3): 1475–76. PMID 18098602. 2009년 3월 26일에 원본 문서 (PDF)에서 보존된 문서. 2019년 2월 18일에 확인함.
- ↑ Friedkin M, Lehninger AL (1949년 4월 1일). “Esterification of inorganic phosphate coupled to electron transport between dihydrodiphosphopyridine nucleotide and oxygen”. 《J. Biol. Chem.》 178 (2): 611–23. PMID 18116985.
- ↑ Preiss J, Handler P (1958). “Biosynthesis of diphosphopyridine nucleotide. I. Identification of intermediates”. 《J. Biol. Chem.》 233 (2): 488–92. PMID 13563526. 2007년 12월 14일에 원본 문서에서 보존된 문서. 2019년 2월 18일에 확인함.
- ↑ Preiss J, Handler P (1958). “Biosynthesis of diphosphopyridine nucleotide. II. Enzymatic aspects”. 《J. Biol. Chem.》 233 (2): 493–500. PMID 13563527.
- ↑ Bieganowski, P; Brenner, C (2004). “Discoveries of Nicotinamide Riboside as a Nutrient and Conserved NRK Genes Establish a Preiss-Handler Independent Route to NAD+ in Fungi and Humans”. 《Cell》 117 (4): 495–502. doi:10.1016/S0092-8674(04)00416-7. PMID 15137942.
- ↑ Chambon P, Weill JD, Mandel P (1963). “Nicotinamide mononucleotide activation of new DNA-dependent polyadenylic acid synthesizing nuclear enzyme”. 《Biochem. Biophys. Res. Commun.》 11: 39–43. doi:10.1016/0006-291X(63)90024-X. PMID 14019961.
- ↑ Clapper DL, Walseth TF, Dargie PJ, Lee HC (1987년 7월 15일). “Pyridine nucleotide metabolites stimulate calcium release from sea urchin egg microsomes desensitized to inositol trisphosphate”. 《J. Biol. Chem.》 262 (20): 9561–8. PMID 3496336. 2007년 12월 14일에 원본 문서에서 보존된 문서. 2019년 2월 18일에 확인함.
- ↑ Imai S, Armstrong CM, Kaeberlein M, Guarente L (2000). “Transcriptional silencing and longevity protein Sir2 is an NAD-dependent histone deacetylase”. 《Nature》 403 (6771): 795–800. Bibcode:2000Natur.403..795I. doi:10.1038/35001622. PMID 10693811.
참고 도서
편집기능
편집- Nelson DL; Cox MM (2004). 《Lehninger Principles of Biochemistry》 4판. W. H. Freeman. ISBN 0-7167-4339-6.
- Bugg T (2004). 《Introduction to Enzyme and Coenzyme Chemistry》 2판. Blackwell Publishing Limited. ISBN 1-4051-1452-5.
- Lee HC (2002). 《Cyclic ADP-Ribose and NAADP: Structure, Metabolism and Functions》. Kluwer Academic Publishers. ISBN 1-4020-7281-3.
- Levine OS, Schuchat A, Schwartz B, Wenger JD, Elliott J (1997). “Generic protocol for population-based surveillance of Haemophilus influenzae type B” (PDF). World Health Organization. Centers for Disease Control. 13쪽. WHO/VRD/GEN/95.05.
- J. Kim, S. H. Lee, F. Tieves, C. E. Paul, F. Hollmann, C. B. Park. "Nicotinamide adenine dinucleotide as a photocatalyst". Science Advances 5, eaax0501 (2019)[1]: 니코틴아마이드 아데닌 다이뉴클레오타이드의 광촉매적 기능
역사
편집- Cornish-Bowden, Athel (1997). 《New Beer in an Old Bottle. Eduard Buchner and the Growth of Biochemical Knowledge.》. Valencia: Universitat de Valencia. ISBN 84-370-3328-4. 2010년 12월 13일에 원본 문서에서 보존된 문서. 2019년 2월 18일에 확인함., A history of early enzymology.
- Williams, Henry Smith (1904). 《Modern Development of the Chemical and Biological Sciences》. A History of Science: in Five Volumes IV. New York: Harper and Brothers., a textbook from the 19th century.
외부 링크
편집- NAD and NADP
- Nicotinamide
- NAD bound to proteins in the Protein Data Bank
- NAD Animation (Flash Required)
- β-Nicotinamide adenine dinucleotide (NAD+ Archived 2013년 10월 19일 - 웨이백 머신 and NADH (reduced) Archived 2013년 10월 19일 - 웨이백 머신 Chemical data sheet from Sigma-Aldrich
- NAD+, NADH and NAD synthesis pathway at the MetaCyc database
- List of oxidoreductases at the SWISS-PROT database
- ↑ Kim, Jinhyun; Lee, Sahng Ha; Tieves, Florian; Paul, Caroline E.; Hollmann, Frank; Park, Chan Beum (2019년 7월 5일). “Nicotinamide adenine dinucleotide as a photocatalyst”. 《Science Advances》 5 (7): eaax0501. doi:10.1126/sciadv.aax0501. 2022년 3월 11일에 확인함.
